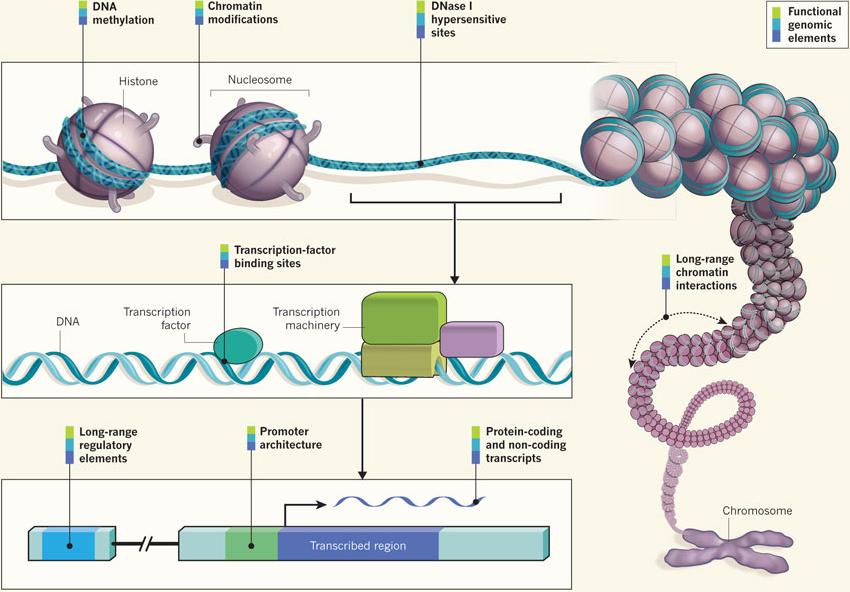
MAPA FUNCIONAL DE GENES QUE SE EXPRESAN EN CÉLULAS HUMANAS
El mapa se ha obtenido con un método basado en el sistema de edición del genoma CRISPR/Cas9 para hacer cambios genéticos en las células.
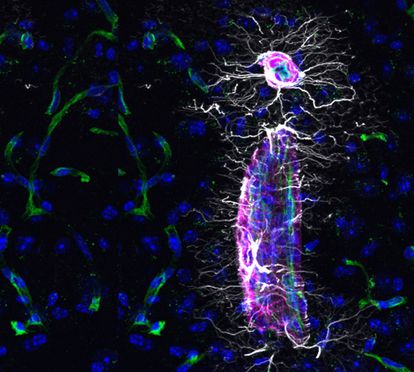
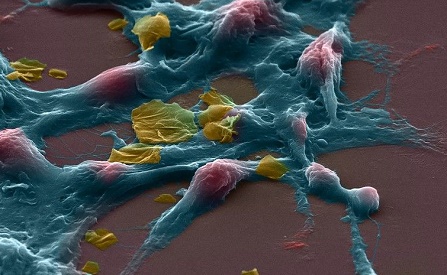
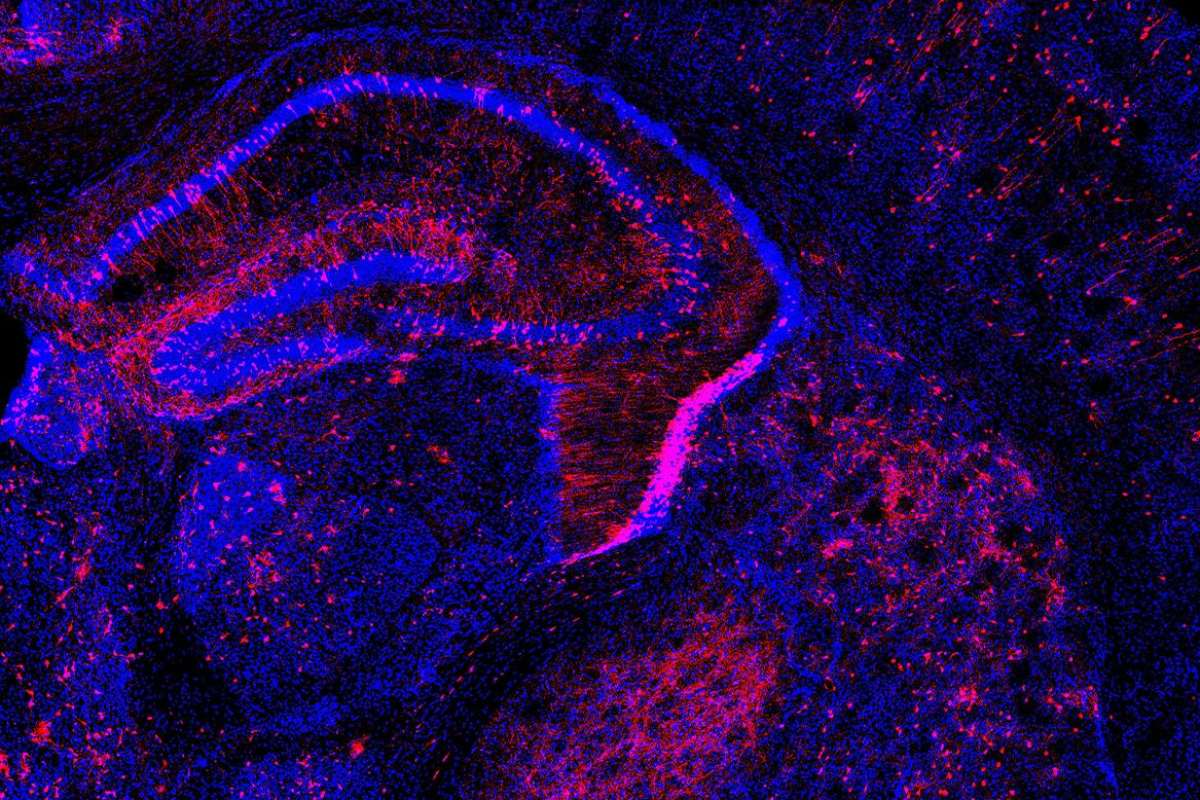 El método ‘Pertub-seq’ permite introducir cambios, como en esta célula de cerebro murino. Foto: LABORATORIO P. ARLOTTA (HARVARD)
El método ‘Pertub-seq’ permite introducir cambios, como en esta célula de cerebro murino. Foto: LABORATORIO P. ARLOTTA (HARVARD)
Investigadores del Instituto Whitehead de Investigación Biomédica del Instituto de Tecnología de Massachusetts (MIT) publican hoy en Cell el primer mapa funcional completo de genes que se expresan en células humanas. Los datos de este proyecto, que relaciona cada gen con su labor en la célula, están disponibles en la página web del laboratorio Weissman para que otros científicos los utilicen. Según sus autores, son la culminación de años de colaboración con el método Perturb-seq (también conocido como CRISP-seq) de secuenciación de una sola célula.
Jonathan Weissman, primer firmante del trabajo, destaca el hecho de que los datos sean accesibles a toda la comunidad científica. «Es un gran recurso en el sentido de que el genoma humano es un gran recurso: puedes acceder e investigar basándote en descubrimientos. En lugar de definir con anticipación qué biología vas a observar, dispones de este mapa de las relaciones genotipo-fenotipo y puedes acceder y revisar la base de datos sin tener que hacer ningún experimento”.
Este abordaje permitió a los investigadores profundizar en diversas cuestiones biológicas: lo usaron para explorar los efectos celulares de genes con funciones desconocidas, para investigar la respuesta de las mitocondrias al estrés y para detectar genes que causan la pérdida o ganancia de cromosomas, un fenotipo que ha resultado difícil de estudiar en el pasado. «Creo que este conjunto de datos permitirá todo tipo de análisis que aún no hemos pensado por parte de personas que provienen de otros campos de la biología y que ahora tienen disponible», ha añadido Tom Norman, ex postdoctorado del laboratorio Weissman y coautor principal del artículo.
El proyecto se basa en Perturb-seq, que permite seguir el impacto de activar o desactivar genes con una profundidad sin precedentes. Este método de secuenciación fue publicado por primera vez en 2016 por un grupo de investigadores que incluye a Weissman y al profesor del MIT Aviv Regev, pero entonces solo podía usarse en pequeños conjuntos de genes y a un gran coste económico. El siguiente paso fue crear una nueva versión de Perturb-seq que pudiera ampliarse. Los resultados se publicaron en una prueba de concepto en Nature Biotechnology en 2020.
El método Perturb-seq usa la edición del genoma CRISPR/Cas9 para introducir cambios genéticos en las células. Además, usa la secuenciación de ARN de una sola célula para capturar información sobre los ARN que se expresan como resultado de un cambio genético determinado. Los ARN controlan todos los aspectos del comportamiento de las células, por lo que este método puede ayudar a decodificar los muchos efectos celulares de los cambios genéticos.
En el estudio recién publicado, los investigadores ampliaron el método a todo el genoma: utilizaron líneas celulares humanas de cáncer de sangre y células no cancerosas derivadas de la retina, realizando el análisis con Perturb-seq en más de 2,5 millones de células y usando los datos para construir un mapa completo que relaciona genotipos con fenotipos.
Al completar el análisis, los investigadores decidieron usar el nuevo conjunto de datos y examinar algunas preguntas biológicas. “La ventaja de Perturb-seq es que te permite obtener un gran conjunto de datos sin sesgos”, ha destacado Tom Norman. “El primer uso fue investigar los genes con funciones desconocidas. Debido a que el análisis también lee los fenotipos de muchos genes conocidos, los investigadores podrían usar los datos para comparar genes desconocidos con los conocidos y buscar resultados transcripcionales similares, lo que podría sugerir que los productos genéticos trabajaron juntos como parte de un complejo más grande.
En el análisis, destacó la mutación del gen C7orf26: los investigadores notaron que los genes cuya eliminación conducía a un fenotipo similar formaban parte de un complejo de proteínas llamado Integrator, que desempeñaba un papel en la creación de pequeños ARN nucleares. El complejo Integrator está formado por muchas subunidades más pequeñas -estudios previos habían sugerido 14 proteínas individuales- y los investigadores pudieron confirmar que C7orf26 constituía un decimoquinto componente del complejo. También descubrieron que las 15 subunidades trabajaban juntas en módulos más pequeños para realizar funciones específicas dentro del complejo Integrator.
Otra ventaja de Perturb-seq es que, debido a que el ensayo se enfoca en células individuales, los investigadores podrían usar los datos para observar fenotipos más complejos que se enturbian cuando se estudian junto con datos de otras células. «A menudo tomamos todas las células en las que se silencia un determinado gen y hacemos una media para ver cómo cambiaron», ha explicado Weissman. “Pero a veces, cuando silencias un gen, diferentes células que están perdiendo ese mismo gen se comportan de manera diferente. Y ese comportamiento puede pasar desapercibido por el estudio conjunto de células”.
Los investigadores encontraron que un subconjunto de genes cuya eliminación condujo a diferentes resultados de una célula a otra era responsable de la segregación cromosómica. Su eliminación estaba causando que las células perdieran un cromosoma o añadieran uno extra (aneuploidia). «No se podía predecir cuál era la respuesta transcripcional a la pérdida de este gen, porque dependía del efecto secundario de qué cromosoma se ganaba o se perdía», ha detallado Weissman. “Nos dimos cuenta de que podíamos cambiarlo y crear este fenotipo compuesto en busca de firmas de cromosomas que se ganaban y perdían. De esta forma, hemos realizado el primer análisis de todo el genoma de los factores necesarios para la correcta segregación del ADN”, ha destacado.
Para Norman, el estudio de la aneuploidía es la aplicación más interesante de estos datos hasta el momento. El motivo es que permite capturar un fenotipo “que solo puedes obtener usando una lectura de una sola célula”.
Los investigadores también utilizaron su conjunto de datos para estudiar cómo respondían las mitocondrias al estrés: cuando alteraron diferentes genes relacionados con las mitocondrias, el genoma nuclear respondió de manera similar a muchos cambios genéticos diferentes. Sin embargo, las respuestas del genoma mitocondrial fueron mucho más variables.
“Todavía hay una pregunta pendiente sobre por qué las mitocondrias todavía tienen su propio ADN”, ha apuntado Joseph Replogle, investigador en el laboratorio Weissman y primer co-autor del artículo. “Una conclusión general de nuestro trabajo es que uno de los beneficios de tener un genoma mitocondrial separado podría ser tener una regulación genética localizada o muy específica en respuesta a diferentes factores estresantes”, ha resumido.
En el futuro, los investigadores esperan usar Perturb-seq en diferentes tipos de células además de la línea de células cancerosas en la que comenzaron su investigación. También esperan continuar explorando su mapa de funciones genéticas y desean que otros hagan lo mismo. “Esta es realmente la culminación de muchos años de trabajo de los autores y otros colaboradores y estoy muy complacido de ver que continúa teniendo éxito y expandiéndose”,.
Referencias
Instituto Whitehead de Investigación Biomédica del Instituto de Tecnología de Massachusetts (MIT) en Cell el primer mapa funcional completo de genes
Javier Granda Revilla. Madrid
Vie, 10/06/2022 – 09:29
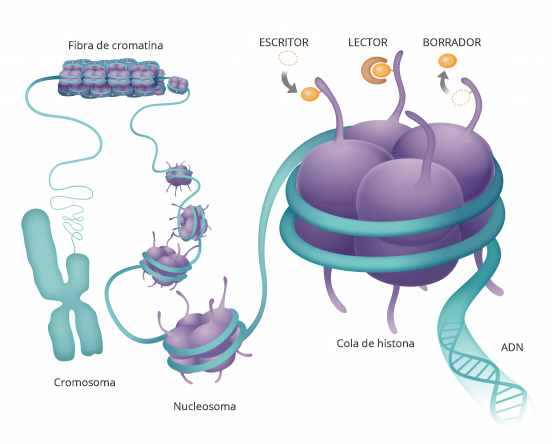
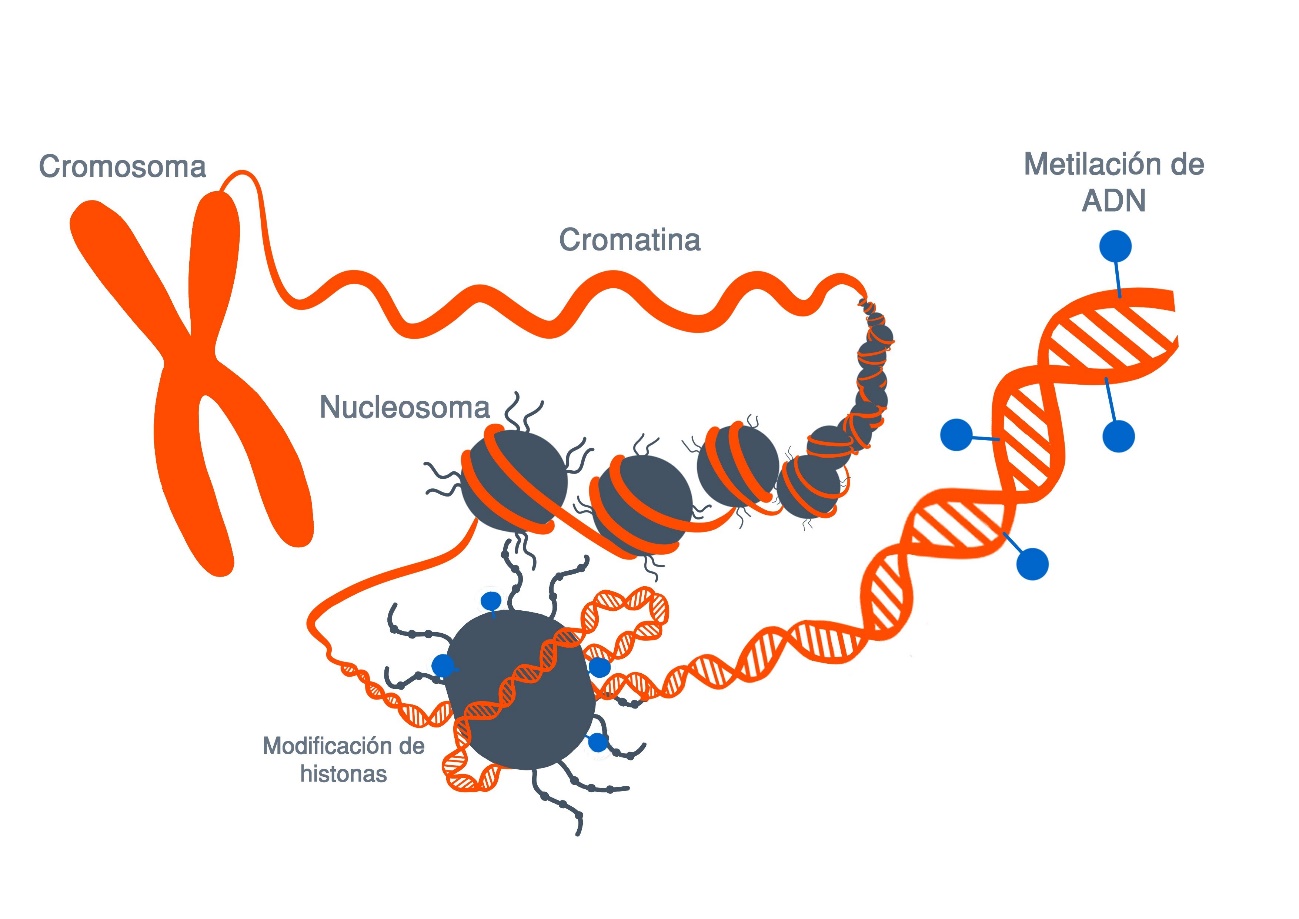
 Metilacion en el dolor
Metilacion en el dolor