La complejidad de la molecula de ADN no tienen limites

Los trabajos de los premios Nobel de química del 2015, sin duda han conformado gran parte de esta misión tan compleja de nuestro ADN
Tomas LindahlU, Paul Modrich y Aziz Sancar han ganado el Nobel han mapeado a nivel molecular cómo las células reparan el ADN dañado para salvaguardar la información genética con su trabajo información fundamental sobre el funcionamiento de las células, un conocimiento que puede ser usado ampliamente y sobre todo sobre el cáncer.
Cada día el ADN es dañado por radiaciones externas e intervienen profundamente en la enfermedad a través de los defectos que se originan en el ADN
Radiaciones ultravioletas, radicales libres u otros agentes cancerígenos, pero a pesar de esos ataques sus moléculas se mantienen intrínsecamente estables. Si el material genético no se desintegra en un completo caos es por la existencia de sistemas moleculares que de forma continua controlan y reparan el ADN. Sancar ha centrado sus trabajos en los sistemas de reparación por escisión de nucleótidos, un mecanismo que subsana los daños causados por las radiaciones ultravioletas. Modrich, por su parte, ha demostrado cómo las células corrigen errores que ocurren cuando el ADN se replica durante la división celular; una variante hereditaria del cáncer de colon, por ejemplo, se debe a un defecto congénito en ese mecanismo.
Cuando éramos estudiantes nos sorprendía la complejidad del ADN, pero no imaginábamos que con el progreso esta complejidad se quedaría pequeña al aparecer los mecanismos de reparación del mismo.
El ADN basura que tanto nos sorprendia es el artesano que repara las alteraciones que en la multiplicación celular ocurren tantas veces en cada dia
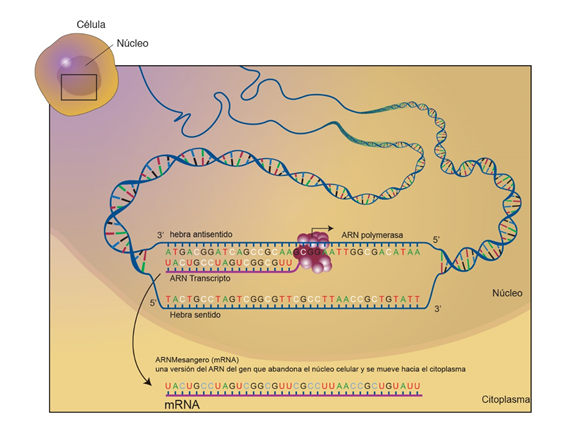
La reparación del ADN es un conjunto de procesos por los cuales una célula identifica y corrige daños hechos a las moléculas de ADN que codifican el genoma. En las células humanas, tanto las actividades metabólicas como los factores ambientales, como los rayos UV o la radiactividad, pueden causar daños al ADN, provocando hasta un millón de lesiones moleculares por célula por día.1 Muchas de estas lesiones causan daños estructurales a la molécula de ADN, y pueden alterar o eliminar la capacidad de la célula de transcribir el gen que codifica el ADN afectado. Otras lesiones producen mutaciones potencialmente nocivas en el genoma de la célula, lo que afecta la supervivencia de sus «células hijas» a la hora de la mitosis. Por consiguiente, el proceso de reparación del ADN es constantemente activo, respondiendo a daños a la estructura del ADN.23
La velocidad de la reparación del ADN depende de muchos factores, como el tipo de célula, su edad, y el ambiente extracelular. Una célula que haya acumulado una gran cantidad de daños en el ADN, o que no pueda reparar eficazmente los daños producidos en su ADN, puede entrar en uno de tres estados posibles:
Un estado irreversible de inactividad, llamado senescencia.
Suicidio celular, llamado apoptosis o muerte celular programada.
Carcinogénesis, o formación de cáncer.
La capacidad de reparación del ADN es vital para la integridad de su genoma, y por tanto, de su funcionamiento normal y el del organismo. En el caso de muchos de los genes que se había demostrado que influían en la longevidad, más tarde se ha revelado que tienen un papel en la reparación y protección del ADN.4 La incapacidad de corregir lesiones moleculares en las células que forman gametos pueden introducir mutaciones en el genoma de sus descendientes, influyendo en el ritmo de la evolución.
Los daños al ADN, que se deben a factores ambientales y a los procesos metabólicos habituales dentro de la célula, tienen lugar a un ritmo de entre mil y un millón de lesiones moleculares por célula por día.1 Aunque esto sólo representa un 0,000165% de las aproximadamente seis mil millones de bases (tres mil millones de pares de bases) del genoma humano, las lesiones no reparadas en genes críticos (como los genes supresores de tumores) pueden impedir que una célula lleve a cabo su función y aumentar de manera significativa la posibilidad de que se forme un tumor.
La inmensa mayoría de los daños al ADN afectan a la estructura primaria de la doble hélice, es decir, la química de las bases mismas es modificada. Estas modificaciones pueden a su vez destruir la estructura normal helicoidal de las moléculas, introduciendo nuevos enlaces químicos o aductos voluminosos que no caben en la doble hélice estándar. A diferencia de las proteínas y el ARN, el ADN suele carecer de estructura terciaria, por lo que no suele haber daños o perturbaciones a este nivel. Sin embargo, el ADN tiene unas superhélices envueltas alrededor de proteínas «empaquetadoras» llamadas histonas (en las eucariotas), y ambas estructuras son vulnerables a los efectos de los daños al ADN.
Los daños al ADN se pueden subdividir en dos tipos principales:
Daños endógenos, como ataques por parte de especies reactivas del oxígeno producidas a partir de subproductos metabólicos normales (mutación espontánea), especialmente en el proceso de desaminación oxidativa;
Daños exógenos causados por agentes externos, tales como:
Radiación ultravioleta del sol [UV 200-300nm] u otras frecuencias de radiación, incluyendo los rayos X y los rayos gamma.5
Hidrólisis o disrupción térmica.
Algunas toxinas vegetales.
Mutágenos artificiales, especialmente compuestos aromáticos que actúan como agentes intercalantes del ADN.
Quimioterapia y radioterapia como tratamiento contra el cáncer.
Virus que se integran en el genoma.7
La replicación de ADN dañado antes de que la célula se divida puede provocar la incorporación de bases erróneas ante las dañadas. Las células hijas que heredan estas bases erróneas llevan mutaciones en los que la secuencia de ADN original es irrecuperable (excepto en el raro caso de una reversión de la mutación, o bien más frecuentemente a través de la recombinación genética a pesar de ser igualmente raro).
Hay cuatro tipos principales de daños al ADN debido a procesos celulares endógenos:
Oxidación de las bases (por ejemplo, 8-oxo-7 0.8-dihidroguanina (8-oxoG)) y generación de interrupciones de la cadena de ADN por parte de especies reactivas del oxígeno.
Alquilación de bases (normalmente metilación), como la formación de 7-metilguanina, 1-metiladenina, O6-metilguanina
Hidrólisis de bases, como desaminación, depurinación y depirimidinación.
Malfuncionamiento de bases, debido a errores en la replicación del ADN, en que se inserta una base de ADN errónea en una cadena de ADN en formación, se inserta una base que no es necesaria insertar, o no se inserta una de necesaria.
Los daños causados por agentes exógenos son de muchos tipos.8 Algunos ejemplos son:
La luz UV-B debido a entrecruzamientos con bases adyacentes de citosina y timina, crean dímeros de pirimidina. Esto recibe el nombre de daño directo al ADN.
La luz UV-A provoca la formación de «radicales libres», especialmente si hay crema solar que ha penetrado en la piel. Esto recibe el nombre de daño indirecto al ADN.
La radiación ionizante, como la que causa la desintegración radiactiva o la de los rayos cósmicos, provoca roturas en las cadenas de ADN.
La disrupción térmica a temperaturas elevadas aumenta la velocidad de la despurinización (pérdida de bases de purina del «tronco» del ADN) y las roturas de cadenas sencillas. Por ejemplo, se observa despurinización hidrolítica en los bacteria termófilas, que viven en aguas termales a 85 a 250 ° C.910 En estas especies, la velocidad de despurinización (300 residuos de purina por genoma por generación) es demasiado alta como para que se repare mediante los mecanismos habituales de reparación, de manera que no se puede descartar la posibilidad de una respuesta adaptativa.
Productos químicos industriales, como el cloruro de vinilo o el agua oxigenada, así como compuestos químicos ambientales como los hidrocarburos policíclicos que se encuentran en el humo, el hollín y el alquitrán, provocan una gran diversidad de aductos-etenobases de ADN, bases oxidadas, fosfotriésteros alquilados, y entrecruzamiento del ADN, por citar sólo algunos efectos.
Los daños por radiación UV, la alquilación/metilación, los daños por rayos X y los daños oxidativos son ejemplos de daños inducidos. Los daños espontáneos incluyen la pérdida de una base, la desaminación, plegamientos de los anillos de azúcar, y los desplazamientos de tautómeros.11
Daños al ADN nuclear y al ADN mitocondrial
En las células humanas, y las células eucariotas en general, el ADN se encuentra en dos puntos de la célula: en el núcleo y en las mitocondrias. El ADN nuclear (ADNn) existe en forma de cromatina durante las fases no replicadoras del ciclo celular, y es condensado en estructuras agregadas denominadas cromosomas durante la división celular. En ambos estados, el ADN es altamente compacto y se enrolla alrededor de proteínas en forma de perla, llamadas histonas. Cuando una célula necesita expresar la información genética codificada en su ADNn, se desenrrolla la región cromosómica correspondiente, expresan sus genes, y luego la región vuelve a ser condensada en su forma de reposo. El ADN mitocondrial (ADNmt) se encuentra dentro de las mitocondrias (un tipo de orgánulos), existe en múltiples copias, y está estrechamente asociado con una serie de proteínas para formar un complejo llamado nucleoide. Dentro de las mitocondrias, las especies reactivas del oxígeno (ERE) y los radicales libres, subproductos de la producción constante de adenosín trifosfato (ATP) mediante la fosforilación oxidativa, crean un medio altamente oxidativo que se sabe que daña la ADNmt. Una enzima clave a la hora de compensar la toxicidad de estas especies es la superóxido dismutasa, que está presente tanto en las mitocondrias como en el citoplasma de las células eucariotas.
El envejecimiento, un estado irreversible en el que la célula ya no se divide (mitosis), es una respuesta protectora en el acortamiento de los extremos de los cromosomas (telómeros). Los telómeros son largas regiones de ADN no codificantes repetitivas, que delimitan los cromosomas y que se degradan parcialmente cada vez que una célula se divide (límite de Hayflick).12 En cambio, la quietud es un estado reversible de latencia que no tiene relación con los daños en el genoma (ciclo celular). El envejecimiento de las células puede representar una alternativa funcional a la apoptosis en que la presencia física de una célula es necesaria para el organismo,13 que sirve como mecanismo de «último recurso» para evitar que una célula con el ADN dañado se replique anormalmente en la ausencia de comunicación celular pro-crecimiento. La división celular incontrolada puede provocar la formación de un tumor (cáncer), que es potencialmente letal para el organismo. Por tanto, la inducción del envejecimiento y la apoptosis es considerada parte de la estrategia de protección contra el cáncer.
Es importante distinguir entre los daños en el ADN y las mutaciones, los dos tipos principales de errores en el ADN.14151617 Los daños en el ADN y las mutaciones son fundamentalmente diferentes. Estos daños son en último término anormalidades químicas en la estructura del ADN, como roturas de cadena sencilla y cadena doble, residuos de 8-hidroxideoxiguanosina, y aductos de hidrocarburos aromáticos policíclicos. Determinadas proteínas pueden reconocer estas alteraciones en el ADN, de manera que los pueden reparar si hay disponible información redundante para ser copiada, a partir de la secuencia intacta de la cadena de ADN complementaria que no ha sufrido esta alteración. Si una célula no repara daños en su ADN, puede quedar parada la expresión de un gen.18
En contraste a los daños en el ADN, una mutación es un cambio en la secuencia de bases del ADN, es decir que no se produce ningún cambio que pueda ser reconocido por las proteínas encargadas de la corrección de estas alteraciones, ya que su composición química y estructural es «normal». Las mutaciones provenientes de los errores de síntesis no pueden ser reconocida por las enzimas una vez que el cambio de bases está presente en ambas cadenas del ADN, de manera que las mutaciones resultan indetectables en la corrección y no son reparadas. Las mutaciones son replicadas durante la división celular.19
A nivel celular, las mutaciones pueden provocar cambios en el metabolismo y la proliferación de estas células.20 En el conjunto de células de un organismo, el número de células mutantes aumentará o disminuirá según los efectos de la mutación en la capacidad de la célula para sobrevivir y reproducirse. Aunque son claramente diferentes los unos de los otros, los daños en el ADN y las mutaciones están relacionados, pues los daños en el ADN provocan a menudo errores de síntesis del ADN durante la replicación o la reparación, y estos errores son una causa importante de mutaciones.
Teniendo en cuenta las propiedades de los daños en el ADN y las mutaciones, se puede ver que los daños en el ADN son un problema especial en células que no se dividen o que lo hacen lentamente, pues los daños no reparados tienden a acumularse con el tiempo. Por otra parte, en las células que se dividen rápidamente, los daños en el ADN no reparados que no matan la célula evitando su replicación suelen provocar errores durante la replicación y, por tanto, mutaciones.21
La gran mayoría de mutaciones que no tienen un efecto neutro son deletéreas para la supervivencia de una célula. Así pues, en una población de células de un tejido con células que se replican, las células mutantes tienden a desaparecer. Sin embargo, las pocas mutaciones que ofrecen una ventaja para la proliferación celular tienden a extenderse de manera clónica a expensas de las células vecinas. Esta ventaja para la célula es una desventaja para el organismo en general, pues estas células mutantes proliferan libremente escapando al control del ciclo celular, son las células cancerosas. Aquellos tipos celulares que se dividen más frecuentemente tienden a acumular más fácilmente las mutaciones, ya que una vez ocurridas las mutaciones tardan estas células poco tiempo en replicar el ADN y por tanto la mutación incorporará poco tiempo con la copia inicial de la cadena complementaria. Así pues una vez dividida una de las dos células resultantes de la división habrá «fijado» la variante mutante, siendo más difícil que ocurra la corrección. Así pues, los daños en el ADN en células que se dividen frecuentemente son una causa importante de cáncer,22 pues dan pie a mutaciones. En cambio, los daños en el ADN en células que se dividen poco son probablemente una causa importante del envejecimiento.23
Las células no pueden funcionar si los daños en el ADN corrompen la integridad y accesibilidad de información esencial en el genoma (pero las células permanecen aparentemente funcionales cuando faltan o están dañados genes «no esenciales»). Según el tipo de daños que ha sufrido la estructura de doble hélice del ADN, han evolucionado una variedad de estrategias de reparación que restauran la información perdida. Si es posible, las células utilizan la cadena de ADN complementaria (si no ha sido modificada) o la cromátida hermana como «plantilla» para restaurar la información original. Si no hay ninguna plantilla disponible, las células utilizan como último recurso un sistema de recuperación propenso a los errores conocido como síntesis de translesión.
Los daños al ADN alteran la configuración espacial de la hélice, su topología, y la célula es capaz de detectar estas alteraciones. Una vez que se detectan los daños, unas moléculas específicas reparadoras del ADN se adhieren al punto dañado o cerca de él, induciendo a otras moléculas a adherirse y formar un complejo que permite que tenga lugar la reparación. Los tipos de moléculas implicados y el mecanismo de reparación que se utiliza depende del tipo de daños que haya sufrido el ADN y de la fase del ciclo celular en que se encuentre la célula.
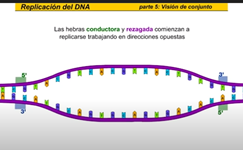
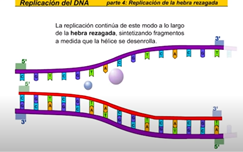
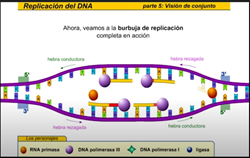
Cuando sólo una de las dos cadenas de la doble hélice tiene un defecto, la otra puede ser utilizada como plantilla para dirigir la corrección de la cadena dañada. Para reparar daños a una de las moléculas pareadas de ADN, existen varios mecanismos de reparación de escisiones, que eliminan el nucleótido dañado y lo sustituyen con un nucleótido intacto complementario al que se encuentra en la cadena de ADN no dañada.
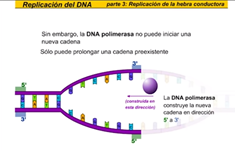
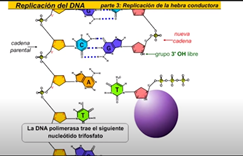
Reparación sobre la marcha, es el principal sistema de corrección de daños. Lo realizan las propias ADN Pol I y ADN Pol III (o sus equivalentes en eucariotas) con su actividad exonucleasa 3′ → 5′ para corregir un nucleótido equivocado que hayan colocado. Esta incorrección es detectada porque el emparejamiento incorrecto causa una distorsión de la doble hélice que las ADN Polimerasas pueden detectar. Sin embargo, la reparación solo puede realizarse si aún no se han puesto más nucleótidos, una vez colocado aunque sea uno más, éste actúa como barrera de no retorno.
Reparación directa, no requiere eliminación de nucleótidos o bases nitrogenadas, sino que se emplean enzimas para reparar directamente alteraciones nucleotídicas. Los principales enzimas empleados son la fotoliasa (separa los dímeros de timinas formados por radiación UV, mediante el mecanismo de fotorreactivación) y la metiltransferasa (retira grupos metilo añadidos al ADN).
Reparación por escisión de base (BER), que repara daños a un único nucleótido causados por oxidación, alquilación, hidrólisis o desaminación. Una glicosidasa escinde la base nitrogenada del nucleótido dañado, generando un sitio apurínico o apirimidínico. El esqueleto pentosa-fosfato residual es eliminado por una AP endonucleasa y finalmente es sustituido por el nucleótido adecuado por la actividad secuencial de ADN polimerasa y ADN ligasa.
Reparación por escisión de nucleótido (NER), que repara daños que afecten cadenas más largas, de entre dos y treinta bases. Este proceso reconoce cambios grandes que distorsionan la hélice, como dímeros de timina, así como roturas de cadena única (reparados con enzimas como la UvrABC endonucleasa. Una forma especializada de NER, conocida como reparación acoplada a transcripción (TCR) desarrolla enzimas de alta prioridad en genes que se están transcribiendo activamente.
Reparación de malapareamiento o reparación por mismatch (MMR). Todas las reparaciones anteriores se realizan antes de terminar la replicación. Este sistema se realiza cuando la replicación ya ha concluido, y corrige errores de nucleótidos mal apareados (pero normales, es decir, no dañados). Para ello debe reconocer qué hebra es la correcta, lo que en procariotas ocurre porque el ADN suele tener metiladas sus bases, pero tras la replicación la hebra nueva no se metila hasta comprobar que no tenga errores, por lo que la maquinaria de reparación supone que si hay un error tras la replicación, se habrá producido en la hebra nueva (la no metilada). Una vez metiladas, o no hay corrección posible, o ésta puede causar errores. Por ejemplo, en cualquier emparejamiento erróneo de GT y CT, se retira preferentemente la timina, porque es probable que sea resultado de la desaminación de la citosina. Este sistema de reconocimiento por metilación solo funciona en procariotas, se ignora cuál es el mecanismo empleado en eucariotas para distinguir la hebra recién formada de la hebra madre.
Estos métodos mencionados hasta ahora reparar el ADN de forma fidedigna, recuperando el genotipo original. Pero cuando los daños son excesivos, se producen los siguientes tipos de reparación, que ya son propensos a errores: no recuperan el genotipo original, se trata de soluciones de emergencia cuando está en juego la supervivencia celular.
Respuesta SOS, que es un rellenado de emergencia que se pone en marcha cuando se acumulan daños que distorsionan la doble hélice (como regiones de ADN monocatenario, por pérdidas de nucleótidos en la cadena complementaria), atascando la maquinaria replicativa. En procariotas esto desencadena el sistema RecA, una proteasa que elimina proteínas represoras de polimerasas de bypass, capaces de sobreponerse a la distorsión de la hélice y rellenar los huecos con nucleótidos al azar. En eucariotas las polimerasas de bypass son constitutivas, están presentes en todo momento en el citoplasma, pero solo se reclutan cuando se acumulan daños, mediante una regulación por ubiquitinación de la abrazadera.
Proteína p53, que más que un sistema de reparación, induce la apoptosis celular cuando los daños no pueden ser reparados ni siquiera por la respuesta SOS, para impedir que se desarrollen tumores.
Las roturas de cadena doble, en el que ambas cadenas de la doble hélice quedan rotas, son especialmente peligrosos para la célula, ya que pueden provocar problemas en el genoma. Existen dos mecanismos que reparan estas roturas: la unión de extremos no homólogos (NHEJ del inglés Non-homologous DNA End Joining) y la reparación recombinativa (también conocida como reparación asistida por plantilla o reparación de recombinación homóloga).
En el NHEJ el ADN ligasa IV, un ADN ligasa especializada que forma un complejo con el cofactor XRCC4, une directamente los dos extremos.24 Para asegurarse de una reparación precisa, el NHEJ se basa en cortas secuencias homólogas llamadas microhomologías, presentes en las colas monocatenarias de los extremos de ADN que deben ser unidos. Si estas secuencias son compatibles, la reparación suele ser correcta.25262728 El NHEJ también puede causar mutaciones durante la reparación. La pérdida de bases nitrogenadas en el lugar de rotura puede provocar deleciones y la unión de translocaciones de forma terminal no correspondientes. El NHEJ es especialmente importante antes de que la célula haya replicado su ADN, pues no hay ninguna plantilla que permita la reparación por recombinación homóloga. Hay rutas de NHEJ «de seguridad» en las eucariotas superiores.29 Además de su papel como «cuidador» del genoma, el NHEJ es necesario para unir roturas de la cadena doble con extremos de horquilla, causados durante la recombinación V (D) J, el proceso que genera la diversidad de los receptores de los linfocitos B y los linfocitos T en el sistema inmunitario de los vertebrados.30
La reparación recombinante requiere la presencia de una secuencia idéntica o casi idéntica que sea utilizada como plantilla para reparar la rotura. La maquinaria enzimática responsable de este proceso es casi idéntica a la maquinaria responsable del cruce cromosómico durante la meiosis. Esta ruta permite que un cromosoma dañado sea reparado utilizando una cromátida hermana (disponible en G2 después de la replicación del ADN) o un cromosoma homólogo como plantilla. Las roturas de cadena doble causados por los intentos de la maquinaria replicante de sintetizar a través de una rotura de cadena única o una lesión no reparada provocan un colapso de la horquilla de replicación y son generalmente reparados por recombinación.
Las topoisomerasas provocan roturas tanto de una única cadena como de la cadena doble cuando cambian el estado de superenrollamiento del ADN, lo que es especialmente habitual en regiones situadas cerca de una horquilla de replicación abierta. Estos roturas no son consideradas como daños en el ADN, ya que son un intermedio natural del mecanismo bioquímico de las topoisomerasas y son inmediatamente reparados por las enzimas que los han creado.
Un grupo de científicos franceses bombardearon Deinococcus radiodurans para estudiar el mecanismo de reparación de roturas de la cadena doble de ADN en este organismo. Al menos dos copias del genoma, con roturas aleatorias del ADN, pueden formar fragmentos de ADN por medio de apareamiento. Entonces, los fragmentos que se solapan parcialmente son utilizados para sintetizar las regiones homólogas mediante un bucle-D en movimiento que puede continuar la extensión hasta que encuentran cadenas correspondientes complementarias. En el último paso se produce un cruce por medio de una recombinación homóloga de recargo dependiente.31
Me impresionan los estudiosos de la bibliografía en general, sin ellos nos relegaríamos a la búsqueda minuciosas y con juchos errores,
Ellos nos lo dan hecho y por ello tienen mi agradecimiento
Referencias
Lodish H., Berk A., Matsudaira P., Kaiser C. A., Krieger M., Scott M. P., Zipursky S. L., Darnell J. (2004). Molecular Biology of the Cell (5ª edición). Nueva York: WH Freeman. p. 963.
Acharya PVN; The isolation and partial characterization of age-correlated oligo-deoxyribo-ribonucleotides with covalently linked aspartyl-glutamyl polypeptides.(June, 1971). Johns Hopkins Med J Suppl, p254-260. PMID 5055816.
Bjorksten, J; Acharya, PVN; Ashman, S; Wetlaufer, DB. Gerogenic Fractions in the Tritiated Rat (July, 1971). Journal of the American Geriatrics Society, p561-574; PMID 5106728.
Browner W. S., Kahn A. J., Ziv E., Reiner A. P., Oshima J., Cawthon R. M., Hsueh W. C., Cummings S. R. (2004). «The genetics of human longevity». Am J Med 117 (11): 851-60.
Hanson Kerry M.; Gratton Enrico; Bardeen Christopher J. (2006). «Sunscreen enhancement of UV-induced reactive oxygen species in the skin». Free Radical Biology and Medicine 41 (8): 1205–1212.
Gulston M, Knowland J. (July 1999). «Illumination of human keratinocytes in the presence of the sunscreen ingredient Padimate-O and through an SPF-15 sunscreen reduces direct photodamage to DNA but increases strand breaks.». Mutat Res. 444 (1): 49–60.
Roulston A., Marcellus R. C., Branton P. E. (1999). «Viruses and apoptosis». Annu. Rev. Microbiol. 53: 577-628. PMID 10547702. doi:10.1146/annurev.micro.53.1.577. Consultado el 20 de diciembre de 2008.
Wolf, FI; Fasanella, S; Tedesco, B; Cavallini, G; Donati, A; Bergamini, E; Cittadini, A. Peripheral lymphocyte 8-OHdG levels correlate with age-associated increase of tissue oxidative DNA damage in Sprague-Dawley rats. Protective effects of caloric restriction. Exp Gerontol, 2005, 40, 181-188.
Madigan MT, Martino JM (2006). Brock Biology of Microorganisms (11th edición). Pearson. p. 136. ISBN 0-13-196893-9.
Toshihiro Ohta, Shin-ichi Tokishita, Kayo Mochizuki, Jun Kawase, Masahide Sakahira and Hideo Yamagata, UV Sensitivity and Mutagenesis of the Extremely Thermophilic Eubacterium Thermus thermophilus HB27, Genes and Environment Vol. 28 (2006), No. 2 p.56–61.
NCBI. DNA Lesions That Require Repair Consultado el 18 de junio de 2010.
Braig M., Schmitt C. A. (2006). «Oncogene-induced senescence: putting the brakes on tumor development». Cancer Res 66: 2881-2884..
Lynch M. D. (2006). «How does cellular senescence prevent cancer?». DNA Cell Biol 25 (2): 69-78.
Armeni, Tatiana; Damiani, Elisabetta; et al. (2004). «Lack of in vitro protection by a common sunscreen ingredient on UVA-induced cytotoxicity in keratinocytes.». Toxicology. 203(1-3) (1-3): 165-178. PMID 15363592. doi:10.1016/j.tox.2004.06.008.
Knowland, John; McKenzie, Edward A.; McHugh, Peter J.; Cridland, Nigel A. (1993). «Sunlight-induced mutagenicity of a common sunscreen ingredient.». FEBS Letters. 324(3): 309-313. doi:10.1016/0014-5793(93)80141-G.
Mosley, C N; Wang, L; Gilley, S; Wang, S; Yu,H (2007). «Light-Induced Cytotoxicity and Genotoxicity of a Sunscreen Agent, 2-Phenylbenzimidazol in Salmonella typhimurium TA 102 and HaCaT Keratinocytes». Internaltional Journal of Environmental Research and Public Health 4 (2): 126-131. doi:10.3390/ijerph2007040006.
Xu, C.; Green, Adele; Parisi, Alfio; Parsons, Peter G (2001). «Photosensitization of the Sunscreen Octyl p-Dimethylaminobenzoate b UVA in Human Melanocytes but not in Keratinocytes.». Photochemistry and Photobiology 73 (6): 600-604. PMID 11421064. doi:10.1562/0031-8655(2001)073<0600:POTSOP>2.0.CO;2.
Bernstein, H; Payne, CM; Bernstein, C; Garewal, H; Dvorak, K. Cancer and aging as consequences of un-repaired DNA damage. In: New Research on DNA Damage (Editor: Frank Columbus) Nova Sci Publ, Hauppauge, NY (n.d.).
Vilenchik, MM; Knudson AG Jr. Inverse radiation dose-rate effects on somatic and germ-line mutations and DNA damage rates. Proc Natl Acad Sci USA, 2000, 97, 5381-5386.
Rutten, BPF; Schmitz, C; Gerlach, OHH; Oyen, HM; de Mesquita, EB; Steinbusch, HWM; Korr, H. The aging brain: accumulation of DNA damage or neuron loss? Neurobiology of Aging, 2007, 28, 91-98.
Linford, NJ; Schriner, SE; Rabinovitch, PS. Oxidative damage and aging: Spotlight on mitochondria. Cancer Res, 2006, 66, 2497-2499. # Liu, Y; Wang, Y; Rusinol, AE; Sinensky, MS; Liu, J; Shell, SM; Zou, Y. Involvement of xerodema pigmentosum group A (XPA) in progeria arising from defective maturation of Prelamin A. FASEB J, 2008, 22, 000-000 (Published on line 9/11/2007)
Gensler, HL; Bernstein, H. DNA damage as the primary cause of aging. Q Rev Biol, 1981, 56, 279-303.
Rao, KS; Loeb, LA. DNA damage and repair in brain: relationship to aging. Mutat. Res, 1992, 275, 317-329.
Wilson T. E., Grawunder U., i Lieber M. R. (1997). «DNA ligase IV mediates non-homologous DNA end joining». Nature 388: 495-498. PMID 9242411.
Moore J. K., Haber J. E. (1996). «Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae». Mol Cell Biol 16 (5): 2164-73. PMID 8628283.
Boulton S. J., Jackson S. P. «Saccharomyces cerevisiae Ku70 potentiates illegitimate DNA double-strand break repair and serves as a barrier to error-prone DNA repair pathways». EMBO J. año = 1996. 15 número = 18: 5093-103. PMID 8890183.
Wilson T. E., i Lieber M. R. (1999). «Efficient processing of DNA ends during yeast nonhomologous end joining. Evidence for a DNA polymerase beta (Pol4)-dependent pathway.». J. Biol. Chem. 274: 23.599-23.609. PMID 10438542.
Budman J, Chu G. (2005). «Processing of DNA for nonhomologous end-joining by cell-free extract.». EMBO J. 24 (4): 849-60. PMID 15692565.
Wang H., Perrault A. R., Takeda Y., Qin W., Wang H., Iliakis G. (2003). «Biochemical evidence for Ku-independent backup pathways of NHEJ». Nucleic Acids Res 31 (18): 5377-88.
Jung D., Alt F. W. (2004). «Unraveling V(D)J recombination; insights into gene regulation». Cell 116 (2): 299-311. PMID 14744439.
Zahradka K., Slade D., Bailone A., Sommer S., Averbeck D., Petranovic M., Lindner A. B., Radman M. (2006). «Reassembly of shattered chromosomes in Deinococcus radiodurans». Nature 443 (7111): 569-573. PMID 17006450. doi:10.1038/nature05160.
Lindahl actualmente es jefe de grupo emérito del Instituto Francis Crick de investigación biomédica en Londres y director emérito de investigación sobre el cáncer en el Laboratorio Clare Hall de Herfordshire (Reino Unido).












 LOS TELÓMEROS
LOS TELÓMEROS