CRISPR-Cas9
El descubrimiento del sistema CRISPR/Cas9 ha revolucionado el campo de la Genómica debido a sus numerosas aplicaciones.
La biología entró en una revolución con el descubrimiento y la aplicación de CRISPR/Cas9 , un conjunto de herramientas moleculares con versatilidad comprobada para realizar modificaciones genéticas con precisión en muchos tipos de células diferentes, incluso en células humanas. Una vez que CRISPR/Cas9 introduce una modificación genética, a menudo se requiere la validación fenotípica a nivel de proteína, y esto se ha logrado tradicionalmente mediante el Western blot. 
ELENA NAVARRO GUERRERO
Sirve para cambiar o «editar» piezas del ADN de una célula. CRISPR-Cas9 utiliza una molécula de ARN con un diseño especial para guiar una enzima, que se llama Cas9, hacia una secuencia particular del ADN. Luego, la Cas9 corta las hebras de ADN en ese lugar y quita una pieza pequeña. Así, se produce un espacio en el ADN en donde se coloca una pieza nueva de ADN. CRISPR-Cas9 es un gran avance en la ciencia que tendrá usos importantes en muchas clases de investigación. En el campo de la investigación del cáncer, puede ayudar a entender cómo se forma el cáncer y cómo responde al tratamiento, así como nuevas formas de diagnosticarlo, tratarlo y prevenirlo.
La ingeniería del genoma utilizando la tecnología CRISPR/Cas9 permite modificaciones genómicas simples, eficientes y precisas en células humanas. Las líneas celulares inmortalizadas convencionales se pueden editar o examinar fácilmente utilizando bibliotecas de todo el genoma con transducción lentiviral. Sin embargo, los tipos de células derivados de la diferenciación de células madre pluripotentes inducidas (iPSC), que a menudo representan modelos derivados de pacientes más relevantes para la patología humana, son mucho más difíciles de diseñar, ya que la entrega de CRISPR/Cas9 a estas células diferenciadas puede ser ineficiente y tóxico. Aquí, presentamos un protocolo de transducción lentiviral eficiente para la entrega de CRISPR/Cas9 a macrófagos derivados de iPSC humana con eficiencias cercanas al 100 %. Demostramos knockouts de CRISPR/Cas9 para tres genes de prueba de concepto no esenciales: HPRT1, PPIB y CDK4. Luego escalamos el protocolo y lo validamos para una pantalla de pérdida de función CRISPR/Cas9 agrupada en todo el genoma. Esta metodología permite, por primera vez, la exploración sistemática de la participación de los macrófagos en las respuestas inmunitarias, la inflamación crónica, las enfermedades neurodegenerativas y la progresión del cáncer, utilizando técnicas eficientes de edición del genoma.
APROVECHAR AL MÁXIMO LOS MACRÓFAGOS
Western blot es una técnica de laboratorio utilizado para detectar una proteína específica en una muestra de sangre o tejido. El método implica el uso de electroforesis en gel para separar las proteínas de la muestra. Las proteínas separadas se transfieren del gel a la superficie de una membrana. La membrana se expone a un anticuerpo específico contra la proteína en estudio. La unión del anticuerpo se detecta usando un marcador radiactivo o químico. Un Western Blot se utiliza a veces para diagnosticar enfermedades.
, Pero cuando se trata de analizar macrófagos derivados de iPSC, el Western blot se queda muy corto «ya que solo teníamos una pequeña cantidad de células porque la línea celular no prolifera». En otras palabras, darse cuenta del emocionante potencial de CRISPR/Cas9 y la transducción lentiviral estuvo cargado de métodos de detección de proteínas obsoletos e ineficientes.
LA SIMPLE SOLUCIÓN OCCIDENTAL PARA PEQUEÑOS TAMAÑOS DE MUESTRA
Al buscar una solución para sus muestras de pequeño tamaño, Elena recurrió a Simple Western, una tecnología de inmunoensayo relativamente nueva que está cobrando impulso para reemplazar el Western blot tradicional para la detección específica de proteínas. Como inmunoensayo automatizado basado en capilares, Simple Western está preparado para reemplazar el Western blot tradicional en parte porque puede analizar tamaños de muestra muy pequeños, tan pequeños como 3 μL, con una sensibilidad a nivel de picogramos. Esto hace posible el análisis de tamaños de muestra pequeños como los macrófagos derivados de iPSC. Como ella describe, «Pude hacer varios análisis [Simple Western] con una muestra pequeña, lo que no podría haber hecho con el Western blot tradicional».
Usando a Wes , un miembro de la familia de instrumentos Simple Western, – Elena Navarro-Guerrero, Ph.D., Senior Postdoctoral Researcher, Nuffield Department of Medicine, Target Discovery Institute podía estar segura de que los golpes de gracia genéticos que hizo con CRISPR/Cas9 y la transducción lentiviral eran reales. En un estudio publicado en 2021 en Scientific Reports , el análisis de Wes mostró la ausencia de los 3 genes no esenciales, HPRT1, PPIB y CDK4, que pretendía eliminar ( FIGURA 1 ). Uno de los resultados significativos de su trabajo es que esta técnica abre la puerta a la exploración sistemática de la participación de los macrófagos en las respuestas inmunitarias, la inflamación crónica, las enfermedades neurodegenerativas y la progresión del cáncer, algo que nunca antes se había hecho.
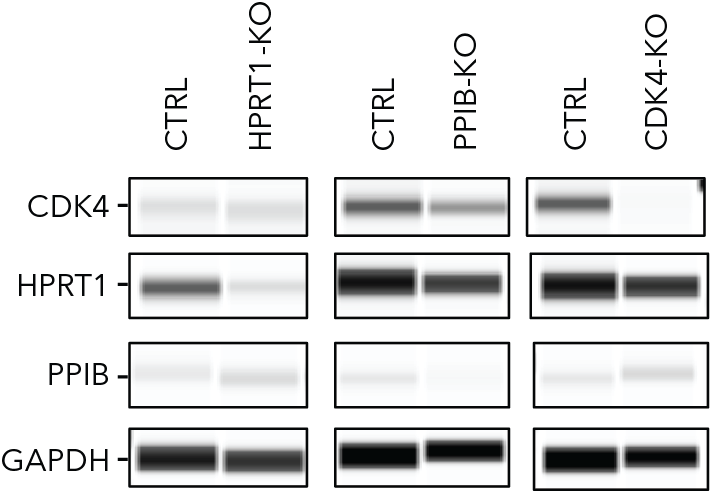
FIGURA 1. Wes confirma la eliminación de 3 genes no esenciales (HPRT1, PPIB y CDK4) en macrófagos derivados de iPSC realizados por CRISPR/Cas9 y transducción lentiviral. Los datos generados por Wes aparecen automáticamente en el software Compass for Simple Western , que controla los sistemas Simple Western. La vista de carril que se muestra aquí fue adaptada con permiso de Navarro-Guerrero et al. (2021) Informes científicos . (CC POR 4.0)
RÁPIDO, CONFIABLE Y MÁS OBJETIVOS POR MUESTRA QUE EL WESTERN BLOT TRADICIONAL
Elena señaló que «Wes es más rápido que el Western blot tradicional». Los resultados se generan en tan solo 3 horas y está totalmente automatizado. Compare esto con el Western blot tradicional, que puede tomar un día completo o más de pasos manuales intensivos, y el ahorro de tiempo se vuelve evidente. A diferencia de los engorrosos pasos del Western blot tradicional que son propensos a errores e introducen variabilidad, Simple Western está completamente automatizado y, por lo tanto, es más confiable y reproducible. Elena también señaló que Simple Western “nos ha permitido analizar varias proteínas en una muestra”. Con este fin, los últimos sistemas Simple Western como Jess y Abby impulsan aún más la detección multiplex con RePlex™, que realiza dos inmunoensayos secuenciales en la muestra en el capilar de muestra. Simple Western y RePlex pueden incluso realizar la detección total de proteínas para normalizar los datos de expresión de proteínas. Si bien la detección de quimioluminiscencia viene de serie con todos los sistemas Simple Western, Jess también incluye dos canales de detección de fluorescencia, lo que impulsa aún más la funcionalidad multiplex.
Referencias
Elena Navarro-Guerrero, Ph.D., Senior Postdoctoral Researcher, Nuffield Department of Medicine, Target Discovery Institute
Sanjana NE, Shalem O, Zhang F. Vectores mejorados y bibliotecas de todo el genoma para la detección de CRISPR. Nat. Métodos. 2014;11(8):783–784. doi: 10.1038/nmeth.3047. –
Horlbeck MA, Gilbert LA, Villalta JE, Adamson B, Pak RA, Chen Y, et al. represión y activación de genes mediada por CRISPR. eLife. 2016;5:e19760. doi: 10.7554/eLife.19760.
Hart T, Tong AHY, Chan K, Van Leeuwen J, Seetharaman A, Aregger M, et al. Evaluación y diseño de pantallas de eliminación de CRISPR/SpCas9 en todo el genoma. G3 Genes Genomas Genet. 2017;7(8):2719.
Parnas O, Jovanovic M, Eisenhaure Thomas M, Herbst Rebecca H, Dixit A, Ye Chun J, et al. Una pantalla CRISPR de todo el genoma en células inmunitarias primarias para diseccionar redes reguladoras. Celúla. 2015;162(3):675–686. doi: 10.1016/j.cell.2015.06.059. –
Ellis EL, Delbrück M. El crecimiento del bacteriófago. J. Gen. Physiol. 1939;22(3):365–384. doi: 10.1085/jgp.22.3.365. –
E. Navarro-Guerrero, C. Tay, J. Whalley, S. Cowley, B. Davies, J. Knight, D. Ebner Eliminación de CRISPR/Cas9 en todo el genoma en macrófagos derivados de células madre pluripotentes inducidas por humanos (iPSC).