La conciencia da significado a lo que somos, lo que queremos, nuestro lugar en el mundo, toda nuestra experiencia.
La conciencia es lo más genuinamente humano, es la interpretación subjetiva de lo que nos pasa. Todo lo que nos sucede viene prendado de señales procedentes de nuestros sentidos y nuestro entorno, pero además viene unido a emociones y recuerdos. Como una especie de voz superpuesta, es uno de los elementos fundamentales de la existencia humana. Nuestra película interna, imprescindible para que la vida tenga sentido y valor. Quizá sea el fenómeno más misterioso del mundo.
Pero aparte de definirla, la conciencia suscita muchas preguntas para las que aún no tenemos buenas respuestas. Se suelen clasificar en fáciles y difíciles. Vayamos con las fáciles: ¿qué tiene de especial el cerebro para generar la conciencia? ¿Está además situada en alguna zona concreta? Y por supuesto, ¿cómo funciona? O, dicho de otra forma, ¿por qué una pieza de materia activa altamente organizada genera una sensación consciente? ¿
¿Dónde está la conciencia?
Un grupo de científicos de la Universidad de Oxford ha descubierto la parte del cerebro donde se encuentra la conciencia humana. … Se trata de la corteza prefrontal lateral del cerebro, el lugar donde, según ellos, se aloja la voz de la conciencia. 2 feb. 2014
SIn embargo la conciencia es mucho mas y esta representada en áreas mas difusas.
Empecemos por los trastornos de conciencia
El Coma
El estado de coma es una alteración grave del nivel de conciencia. En el coma el paciente presenta un estado de completa falta de respuesta (exceptuando únicamente algunos reflejos automáticos).
se pueden identificar diferentes grados de nivel de conciencia, que va desde el estado de alerta normal hasta lo que se conoce como muerte cerebral.
Estado de alerta: situación normal en una persona sana, tanto en vigilia como en sueño fisiológico.
Obnubilación-confusión: cuando existe una disminución moderada del nivel de conciencia, pero el paciente se puede despertar fácilmente con estímulos, hay una alteración de la atención y respuestas lentas. Puede aparecer también desorientación témporo-espacial y agitación (estado confusional), especialmente nocturna, alternando con periodos de lucidez.
Estupor: el paciente está dormido, con un nivel de conciencia muy disminuido, y con mínimas respuestas verbales y motoras a los estímulos.
Coma: estado de falta de respuesta ante cualquier estímulo. El paciente permanece con los ojos cerrados. Durante un estado de coma el tronco encefálico, parte del cerebro que controla las funciones vitales como la respiración, está activo. Así, el organismo es capaz de mantenerse mientras se suplan necesidades básicas como la alimentación. El paciente respira, regula las constantes vitales, pero no hay actividad en las áreas superiores del cerebro ni aparece movimiento alguno que indique un mínimo nivel de conciencia.
Estado vegetativo persistente: estado de falta de conciencia total pero que mantiene apertura espontánea ocular durante periodos de despertar. Los pacientes mantienen la función respiratoria, alternando periodos de sueño aparente con periodos de vigilia con los ojos abiertos. No hay ninguna actividad motora voluntaria
Muerte cerebral: situación de ausencia total de respuesta cerebral que incluye la ausencia de funciones automáticas como la respiratoria. Es una situación irreversible.
Las causas del coma son múltiples, y engloban a todas aquellas que afecten al sistema nervioso central. Ello incluye:
Traumatismos craneoencefálicos.
Tóxicos
Hipoxia (falta de oxígeno por paro cardiaco , por ejemplo)
Lesiones vasculares cerebrales (hemorragia, infarto cerebral)
Infecciones del SNC (meningoencefalitis)
Intoxicaciones o envenenamientos
Tumores cerebrales
Enfermedades metabólicas (encefalopatía mitocondrial)
Trastornos del desarrollo (hidrocefalia congénita)
El diagnóstico se basa en la exploración neurológica, donde mediante una serie de maniobras de estimulación sencilla se puede establecer el grado de nivel de conciencia.
Para conocer la causa el médico puede indicar pruebas como EKG (infarto grave), analítica con tóxicos, pruebas de neuroimagen (lesiones cerebrales). El EEG (registro de la actividad cerebral) es útil durante la evolución del coma para distinguir otros estados como la muerte encefálica donde no existe ningún tipo de trazado eléctrico.
La evolución del coma puede ser hacia un estado vegetativo idéntico al coma pero en el que sí se producen movimientos espontáneos de ojos, o a un estado de mínima conciencia en el que el paciente puede llegar a seguir a alguien con la mirada o responder instrucciones muy básicas, a un empeoramiento y complicaciones que conllevan al fallecimiento o a su recuperación.
En el coma el paciente respira, regula las constantes vitales, pero no hay actividad en las áreas superiores del cerebro ni movimiento alguno que indique un mínimo nivel de conciencia.
El diagnóstico se basa en la exploración neurológica, donde mediante una serie de maniobras de estimulación sencilla se puede establecer el grado de nivel de conciencia.
El pronóstico y evolución de la persona en coma es aún un desafío. Depende sobre todo de la causa, y también de la zona anatómica y extensión de la afectación (daño neurológico) y la edad.
Anatomia de la conciencia
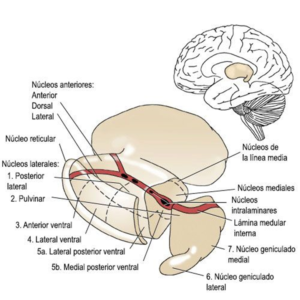
Las estructuras neuroanatómicas encargadas del mantenimiento del nivel de consciencia son múltiples y variadas, interviniendo prácticamente todas las vías y centros nerviosos en mayor o menor medida, los cuales están organizados jerárquicamente y cooperan de forma funcional. Entre ellos el principal sistema encargado es el denominado Sistema Reticular Activador Ascendente (SRAA), definido de forma clásica por los experimentos de Moruzzi y Magoun, como una red neuronal originada en el tegmentum de la protuberancia y mesencéfalo
El sustrato anatómico de la consciencia comprende el SRAA desde el nivel mediopontino hasta los núcleos intralaminares, los de la línea media y los talámicos e hipotalámicos paraventriculares junto a sistemas corticales cerebrales difusamente interconectados
Tálamo y conciencia
Investigadores de EE UU han conseguido estimular el tálamo lateral central de un macaco anestesiado y han encontrado vínculos entre esta región y la capacidad de experimentar sensaciones conscientes. Las técnicas empleadas podrían servir para despertar a la gente que se encuentra en coma. El estado de coma es una alteración grave del nivel de conciencia. Que es el coma, un paciente presenta un estado de completa falta de respuesta (exceptuando únicamente algunos reflejos automáticos).
Se pueden identificar diferentes grados de nivel de conciencia, que va desde el estado de alerta normal hasta lo que se conoce como muerte cerebral.
Estado de alerta: situación normal en una persona sana, tanto en vigilia como en sueño fisiológico.
Obnubilación-confusión: cuando existe una disminución moderada del nivel de conciencia, pero el paciente se puede despertar fácilmente con estímulos, hay una alteración de la atención y respuestas lentas. Puede aparecer también desorientación témporo-espacial y agitación (estado confusional), especialmente nocturna, alternando con periodos de lucidez.
Estupor: el paciente está dormido, con un nivel de conciencia muy disminuido, y con mínimas respuestas verbales y motoras a los estímulos.
Coma: estado de falta de respuesta ante cualquier estímulo. El paciente permanece con los ojos cerrados. Durante un estado de coma el tronco encefálico, parte del cerebro que controla las funciones vitales como la respiración, está activo. Así, el organismo es capaz de mantenerse mientras se suplan necesidades básicas como la alimentación. El paciente respira, regula las constantes vitales, pero no hay actividad en las áreas superiores del cerebro ni aparece movimiento alguno que indique un mínimo nivel de conciencia.
Estado vegetativo persistente: estado de falta de conciencia total pero que mantiene apertura espontánea ocular durante periodos de despertar. Los pacientes mantienen la función respiratoria, alternando periodos de sueño aparente con periodos de vigilia con los ojos abiertos. No hay ninguna actividad motora voluntaria
Muerte cerebral: situación de ausencia total de respuesta cerebral que incluye la ausencia de funciones automáticas como la respiratoria. Es una situación irreversible.
tálamo lateral central tiene una función primordial en la conciencia. / Pixabay
Los expertos en neurociencia llevan años preguntándose en qué parte del cerebro surge la conciencia, es decir, la capacidad de experimentar sensaciones internas y externas. Ahora, un nuevo estudio publicado en la revista Neuron indica que un área específica del cerebro –el tálamo lateral central– parece desempeñar un papel clave.
Los investigadores consiguieron limitar las partes del cerebro que estaban más vinculadas a la conciencia
Estudios previos, incluyendo algunos en humanos, sugerían que ciertas áreas, como la corteza parietal y el tálamo, estaban involucradas en dicha capacidad y destacaban la comunicación ellas. “Decidimos ir más allá del enfoque clásico, que estudiaba la actividad de cada área de una en una”, dice Yuri Saalmann, profesor de la Universidad de Wisconsin-Madison (EE UU) y autor principal. “Grabamos múltiples áreas al mismo tiempo para ver cómo se comportaba toda la red”,.
Según los autores, la corteza cerebral tiene seis capas que desempeñan diferentes papeles en el procesamiento y la comunicación neuronal. “Utilizamos sondas laminares que pueden abarcar las capas corticales y grabar desde todas ellas simultáneamente”, explica a SINC Michele Redinbaugh, estudiante de posgrado en el departamento de psicología de la institución estadounidense y autora del trabajo.
Así consiguieron limitar las partes del cerebro que eran importantes y las vías de comunicación de las capas que estaban más vinculadas a la conciencia. También descartaron otras áreas que anteriormente se habían relacionado con ella.
Animales despiertos de la anestesia
Para llevar a cabo la investigación, los científicos utilizaron macacos como modelo animal y los estudiaron despiertos, dormidos y anestesiados. Estimularon el tálamo lateral central, que se encuentra en el centro del cerebro anterior (prosencéfalo), con unos electrodos más pequeños de lo habitual, diseñados específicamente para esta prueba.
“Nuestros electrodos tienen un diseño muy diferente”, apunta Saalmann. “Están mucho más adaptados a la forma del cerebro que queremos estimular. También imitan con más detalle la actividad eléctrica que se tiene un sistema normal y saludable”, aclara.
“Actuaron como si estuviesen despiertos y, cuando desconectamos la estimulación, los animales volvieron directamente a estar inconscientes”
Estimular esta área fue suficiente para despertar a los animales que estaban anestesiados y provocar comportamientos normales de vigilia. “Cuando estimulábamos esta zona, podíamos despertar a los animales y restablecer toda la actividad neuronal que normalmente tendrían en la corteza cerebral durante la vigilia”, afirma Saalmann. “Actuaron como si estuviesen despiertos y, cuando desconectamos la estimulación, los animales volvieron directamente a estar inconscientes”, concreta.
Acercamiento a los trastornos de la conciencia
Para comprobar el estado de vigilia, se examinó su respuesta neuronal a una estimulación auditiva que consistía en activar una serie de pitidos intercalados con otros sonidos aleatorios. Los animales respondieron de la misma manera en la que los animales despiertos lo harían.
“La motivación primordial de esta investigación es ayudar a las personas con trastornos de la conciencia a vivir mejor”, asegura Redinbaugh. El objetivo de su estudio es entender los mecanismos mínimos del estado de conciencia para dirigir clínicamente la parte correcta del cerebro.
“Es posible que podamos usar este tipo de electrodos estimulantes del cerebro para sacar a la gente del coma. Nuestros hallazgos también pueden ser útiles para desarrollar nuevas formas de monitorear a los pacientes bajo anestesia clínica, para asegurarnos de que estén inconscientes de manera segura”, explica.
El Coma de Pase, es un problema en neurología y los que hemos tenido ocasión de tratar este tipo de pacientes, nos desesperábamos al igual que sus familias. Al ver enfermos en coma, siempre
Referencia bibliográfica:
Redinbaugh, M. et al. “Thalamus Modulates Consciousness via Layer-Specific Control of Cortex”. Neuron. 12 de febrero de
María Marín 12/2/2020 17:00 CEST Dra. Eva Ormaechea
Especialista en Medicina Intensiva
Médico consultor de Advance Medical
 Fuente:
Fuente: