ATLAS CELULAR HUMANO
– USANDO LA TÉCNICA DE SECUENCIACIÓN DE ARN DE CÉLULA ÚNICA
El consorcio internacional desvela el transcriptoma de más de un millón de células individuales de 33 órganos.
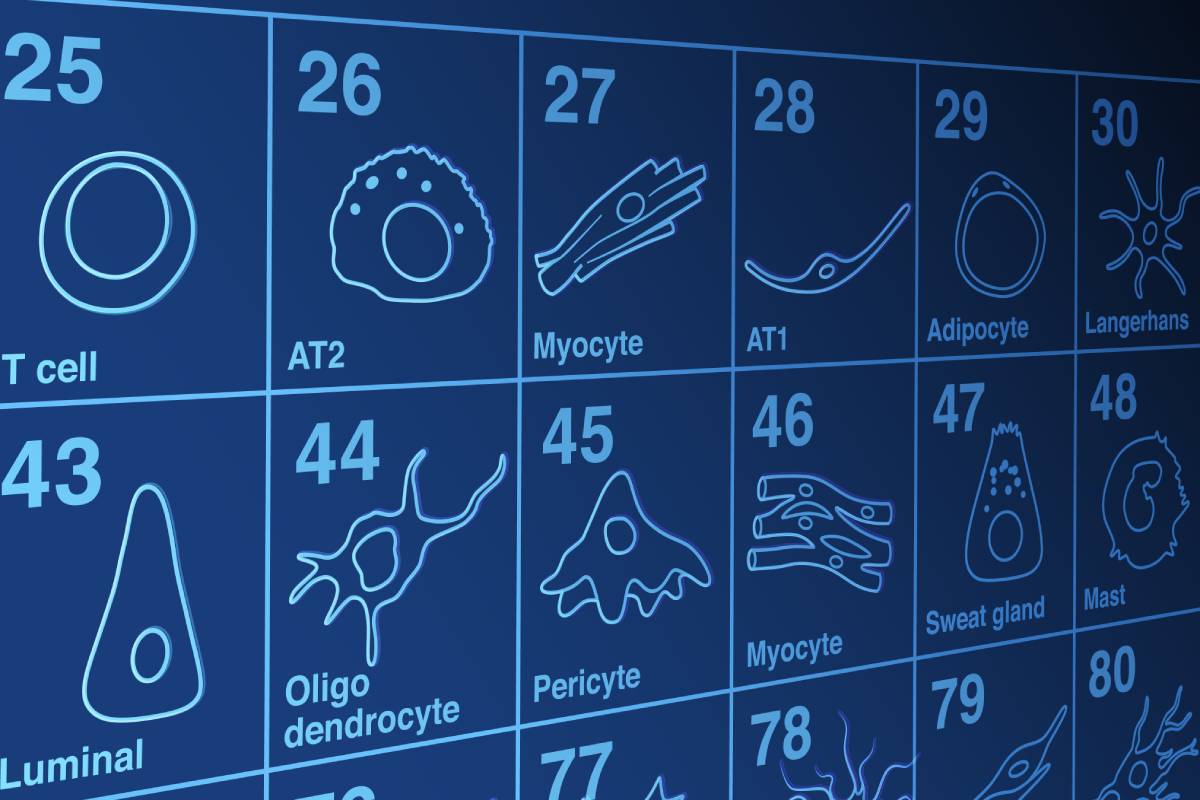 Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas
Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas
Mar de Miguel. Madrid Jue, 12/05/2022 – 21:09
El estudio del cuerpo humano se puede realizar a diferentes niveles. Podemos fijarnos en la anatomía, como se ha hecho a lo largo de los siglos; en las células y tejidos, como se avanzó con el descubrimiento del microscopio; o en sus cromosomas y genes, como se ha conseguido con la secuenciación de nuestro genoma. Después, podemos aplicar distintos enfoques. Mientras que la biología y la bioquímica intentan explicar procesos, la medicina se centra en las enfermedades para curarlas.
Con el avance de la ciencia, cada vez que se descubre algo, podemos profundizar en lo que ya sabíamos o combinar nuestros métodos para multiplicar los hallazgos. Esto lo que ocurre con las nuevas herramientas de secuenciación. El genoma que hemos obtenido para nuestra especie no es el de un humano o un órgano en concreto, sino que es la secuencia completa de un modelo ideal, en general. Aún falta por averiguar algo importante. ¿Cómo es nuestro genoma célula a célula?
La técnica que lo hace posible es la secuenciación de célula única (scRNA-seq, siglas en inglés de single cell RNA sequencing, basada en la tecnología del ARN). Hablamos de la secuencia de una sola célula, lo que abre todo un abanico de posibilidades. Con ella, los científicos cartografían el transcriptoma (de cualquier especie y de cualquier órgano) a nivel unicelular, con todas las diferencias que hay entre las células, lo que también incluye sus diferentes estadios. La información que se obtiene de estos atlas celulares es colosal y se organiza en una base de datos para cada célula.
Sus aplicaciones se disparan. En embriología, sirven para identificar de dónde procede cada célula. En el desarrollo, para trazar los linajes celulares después de nacer, los de cualquier célula en división y sus derivadas. En evolución, para comparar las células homólogas de diferentes especies. En medicina y farmacia, para identificar el origen de una enfermedad, desarrollar métodos de diagnóstico aún más finos, idear nuevos tratamientos o evaluar la eficacia de un fármaco.
 Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas
Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas
Este jueves, la revista Science ha publicado un importante avance en este campo, al presentar, con cuatro nuevas publicaciones, el mayor Atlas Celular Humano hasta la fecha. No es la primera vez que se secuencian nuestros órganos célula a célula. Esta línea de investigación es de tales dimensiones que quien dirige los trabajos, el Consorcio internacional Human Cell Atlas (HCA), ya había desvelado los atlas celulares de distintos órganos, como el del cerebro, pulmón o riñón.
Hasta la fecha, se habían centrado en órganos y tejidos de manera individual. Ahora, llega una serie de mapas transversales, los llamados mapas entre tejidos, el atlas de más de un millón de células individuales de 33 órganos. Se trata de un descubrimiento de grandes implicaciones terapéuticas, tanto para enfermedades comunes como raras, para el desarrollo de vacunas, la inmunología antitumoral o la medicina regenerativa.
Cuatro publicaciones simultáneas
Cuando se trata de grandes descubrimientos las revistas científicas sincronizan su publicación a la vez. Esto es lo que ha ocurrido con este nuevo atlas celular y sus cuatro artículos correspondientes.
En el primer estudio, se presenta la Tabula Sapiens, un atlas celular que aporta 400 tipos de células diferentes obtenidos de 24 órganos. Para ensamblar los datos, se ha secuenciado el ARN de 500.000 células vivas de distintas personas, analizando las que venían de un solo donante y haciendo comparaciones cruzadas controladas por sus antecedentes genéticos, edad, exposición ambiental y efectos epigenéticos.
El segundo atlas se ha hecho a partir de más de 200.000 células congeladas. Las muestras proceden de 8 órganos humanos sanos de 16 donantes. Además, se han empleado técnicas de aprendizaje automático para asociar las células del atlas con miles de enfermedades relacionadas con un solo gen, así como dolencias relativas a factores genéticos complejos. El objetivo es descubrir qué tipos de células y qué genes están involucrados en ciertas enfermedades.
El sistema inmunitario humano se ha abordado en el tercer estudio, que ofrece el atlas celular del sistema inmunitario, no solo de células sanguíneas, sino de las células que están localizadas en el resto de los tejidos. Para ello, los autores de este equipo de investigación han secuenciado células inmunitarias adultas y en desarrollo de 16 tejidos de 12 donantes adultos, obteniendo la expresión génica de más de 300.000 células. Además, han desarrollado una herramienta de inteligencia artificial llamada CellTypist con la que han identificado 101 tipos celulares distintos, bien por sus características o por su estado, analizando más de un millón de células.Por último, el cuarto artículo, se ha centrado en 9 tejidos prenatales. Así, se ha elaborado un atlas unicelular y espacial del sistema inmunitario en todas las etapas de gestación. Este trabajo revela que el desarrollo de las células sanguíneas y las del sistema inmune no solo se produce en los órganos hematopoyéticos primarios sino en muchos tejidos periféricos.
Del transcriptoma a la información espacial
Como estos atlas celulares se basan en información genómica, no todos ellos incluyen información sobre la morfología de las células que se analizan, es decir, no siempre vemos cómo son esas células o dónde se sitúan. Para ello, es preciso combinar otros métodos, como los análisis histológicos o los mapas celulares de alta resolución.
“En nuestro estudio sobre el desarrollo del sistema inmunológico humano sí hemos incluido una imagen de campo claro bajo la información transcriptómica, por lo que tenemos la información morfológica de las células y del microambiente del tejido en el que se encuentran”, ha afirmado a Diario Médico Sarah Teichmann, directora de Genética Celular del Instituto Wellcome Sanger y copresidenta del comité organizador del Atlas Celular Humano. “La resolución es de 50 micras, por lo que no se trata de una sola célula precisamente. El incluir otras tecnologías de mapeo está definitivamente en el radar de la comunidad científica”, ha asegurado.
Para Stephen Quake, presidente de CZ Biohub Network y catedrático de bioingeniería y de física aplicada de la Universidad de Stanford, aún se puede hacer más. “Muchos de nuestros tejidos los han analizado patólogos capacitados. Tenemos tinciones e imágenes ópticas que proporcionan una base para empezar a conectar la morfológica con el perfil de expresión génica. Además, solo usamos una pequeña fracción de todos los tejidos para hacer el trabajo, por lo que podemos volver atrás y revisarlos”, ha comentado Quake a este diario.
“En los atlas celulares tenemos dos brazos, el brazo celular y el brazo espacial”, ha explicado Aviv Regev, copresidenta del Atlas Celular Humano y catedrática Instituto Broad y del MIT. “El plan es tener ambos totalmente representados. También quiero destacar la importancia de los algoritmos de aprendizaje profundo, que nos ayudan a combinar un mismo tipo de datos de diferentes tejidos y son excelentes para identificar si lo que parecen manzanas o naranjas, en realidad, son la misma cosa”, ha añadido.
Secuenciada por primera vez la ‘zona gris’ del genoma humano
Un estudio en organoides muestra que los diabéticos son más susceptibles al SARS-CoV-2
Así identifica el cerebro que hemos cometido un error y aprendemos
Uno de los pioneros y mayores expertos del mundo en la técnica de secuenciación de una única célula dirige varios de los proyectos para la construcción del atlas celular humano. Se trata de Kun Zhang, catedrático del Departamento de Bioingeniería de la Universidad de California, San Diego (UCSD). Zhang ha participado en el encuentro Age Open Science que se ha celebrado esta semana en Salamanca y al que ha acudido Diario Médico.
Allí ha comparado el esfuerzo científico en obtener el atlas celular humano con la secuenciación de nuestro genoma. “Hace veinte años, cuando se elaboró el gran proyecto del genoma humano, hubo varios grupos que trabajaron solo en uno o dos cromosomas, como el cromosoma 21 o el 13. Cuando lo juntaron todo, se obtuvo el gran genoma humano”, ha explicado a este medio.
Aunque, en esta ocasión, Zhang no forma parte del macroestudio publicado este jueves en Science, sí lo ha hecho con anterioridad. “Esta investigación la coordina un gran consorcio para el que mi grupo ha contribuido con el atlas de varios órganos humanos: el atlas del cerebro, del pulmón y del riñón”, ha señalado Zhang. “Las publicaciones que salen ahora van a tener un significado importante», ha afirmado. “Serán como cuando se trabajó en uno de esos cromosomas hace 20 años, cuando se trataba de publicar el genoma humano”.
El de la especie humana no es solo el único atlas que se ha construido. Los hay de muchos otros organismos, de distintos órganos o tejidos. El de primates no humanos (macacos) se ha publicado recientemente en la revista Nature. En él han participado 35 instituciones internacionales. Entre ellas está el grupo del CIBER de Enfermedades Neurodegenerativas liderado por Pura Muñoz, la Premio Nacional de Investigación Ramón y Cajal de Biología 2021, profesora de Biología Celular ICREA de la Universidad Pompeu Fabra (UPF) e investigadora del laboratorio de regeneración tisular del Centro Nacional de Investigaciones Cardiovasculares (CNIC).
Células senescentes
Muñoz, que no ha participado en el atlas celular humano, es experta en los procesos de envejecimiento y, gracias a la técnica de scRNA-seq ha identificado distintos tipos de células senescentes, como ha expuesto en el encuentro de Salamanca. Desde allí, ha valorado el estudio en humanos para este diario y ha comentado la repercusión que los atlas celulares tendrá en el estudio de enfermedades relacionadas con la edad.
“Las células senescentes forman parte de los tejidos cuando envejecemos. También están presentes siempre que haya un daño”, ha comentado. El proceso de daño tisular conlleva la aparición de estas células, “que algunas veces son beneficiosas, pero, casi siempre son perjudiciales”, ha indicado. Lo que ocurre es que, al envejecer, comienzan a fallar los mecanismos que se encargan de eliminarlas y, con el paso del tiempo, “ejercen un papel negativo sobre las células de su entorno. Son unos invitados que causan problemas cuando hay daño y el problema es que aumentan durante el envejecimiento”, ha asegurado Muñoz.
En cuanto a las técnicas de secuenciación de célula única, tanto del primate como del ser humano “vamos a ver que aumenta el número de células dañadas, que no son buenas, en los tejidos”, ha destacado Muñoz. “Estas publicaciones nos dicen cómo estamos cuando envejecemos para poder ver las causas del envejecimiento y ver qué podemos hacer para revertirlo. Si no conocemos bien cómo estamos, no podemos hacer nada para luchar contra ello”, ha añadido.
ATLAS CELULAR HUMANO GENÉTICA
– USANDO LA TÉCNICA DE SECUENCIACIÓN DE ARN DE CÉLULA ÚNICA
El consorcio internacional desvela el transcriptoma de más de un millón de células individuales de 33 órganos.
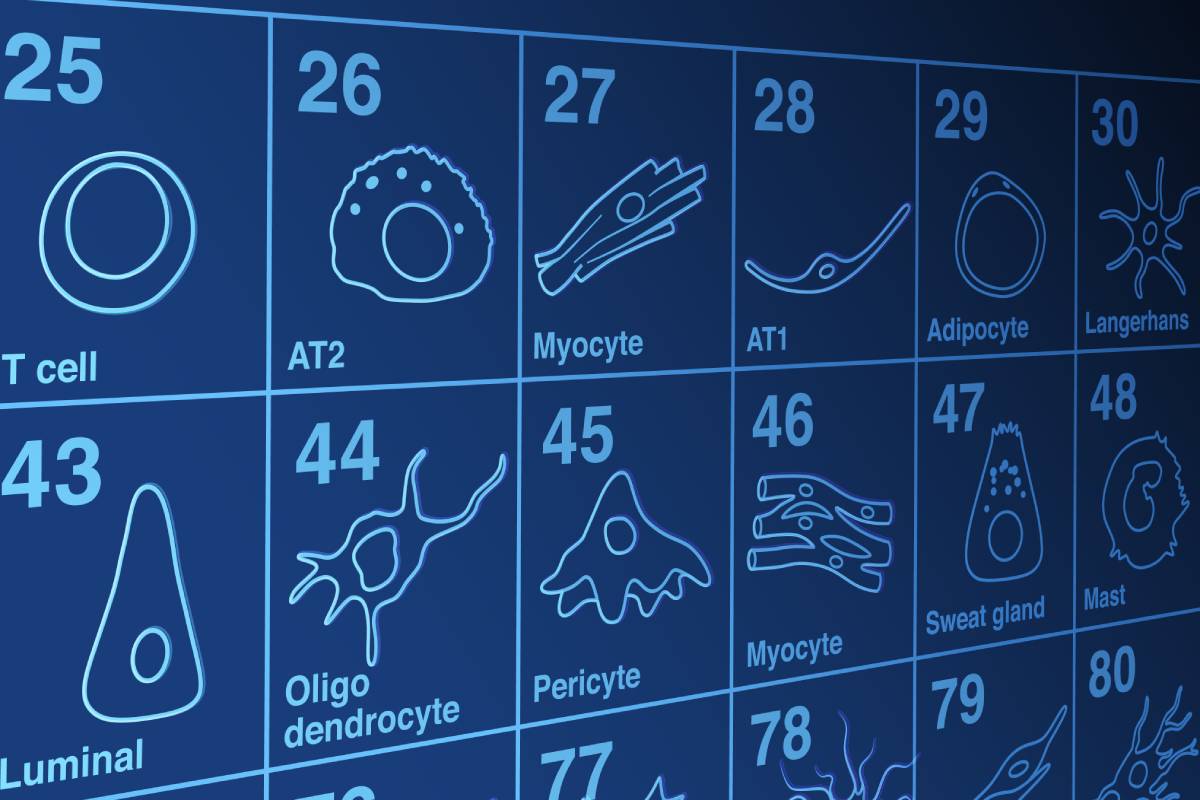 Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas
Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas
Mar de Miguel. Madrid Jue, 12/05/2022 – 21:09
El estudio del cuerpo humano se puede realizar a diferentes niveles. Podemos fijarnos en la anatomía, como se ha hecho a lo largo de los siglos; en las células y tejidos, como se avanzó con el descubrimiento del microscopio; o en sus cromosomas y genes, como se ha conseguido con la secuenciación de nuestro genoma. Después, podemos aplicar distintos enfoques. Mientras que la biología y la bioquímica intentan explicar procesos, la medicina se centra en las enfermedades para curarlas.
Con el avance de la ciencia, cada vez que se descubre algo, podemos profundizar en lo que ya sabíamos o combinar nuestros métodos para multiplicar los hallazgos. Esto lo que ocurre con las nuevas herramientas de secuenciación. El genoma que hemos obtenido para nuestra especie no es el de un humano o un órgano en concreto, sino que es la secuencia completa de un modelo ideal, en general. Aún falta por averiguar algo importante. ¿Cómo es nuestro genoma célula a célula?
La técnica que lo hace posible es la secuenciación de célula única (scRNA-seq, siglas en inglés de single cell RNA sequencing, basada en la tecnología del ARN). Hablamos de la secuencia de una sola célula, lo que abre todo un abanico de posibilidades. Con ella, los científicos cartografían el transcriptoma (de cualquier especie y de cualquier órgano) a nivel unicelular, con todas las diferencias que hay entre las células, lo que también incluye sus diferentes estadios. La información que se obtiene de estos atlas celulares es colosal y se organiza en una base de datos para cada célula.
Sus aplicaciones se disparan. En embriología, sirven para identificar de dónde procede cada célula. En el desarrollo, para trazar los linajes celulares después de nacer, los de cualquier célula en división y sus derivadas. En evolución, para comparar las células homólogas de diferentes especies. En medicina y farmacia, para identificar el origen de una enfermedad, desarrollar métodos de diagnóstico aún más finos, idear nuevos tratamientos o evaluar la eficacia de un fármaco.
 Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas
Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas
Este jueves, la revista Science ha publicado un importante avance en este campo, al presentar, con cuatro nuevas publicaciones, el mayor Atlas Celular Humano hasta la fecha. No es la primera vez que se secuencian nuestros órganos célula a célula. Esta línea de investigación es de tales dimensiones que quien dirige los trabajos, el Consorcio internacional Human Cell Atlas (HCA), ya había desvelado los atlas celulares de distintos órganos, como el del cerebro, pulmón o riñón.
Hasta la fecha, se habían centrado en órganos y tejidos de manera individual. Ahora, llega una serie de mapas transversales, los llamados mapas entre tejidos, el atlas de más de un millón de células individuales de 33 órganos. Se trata de un descubrimiento de grandes implicaciones terapéuticas, tanto para enfermedades comunes como raras, para el desarrollo de vacunas, la inmunología antitumoral o la medicina regenerativa.
Cuatro publicaciones simultáneas
Cuando se trata de grandes descubrimientos las revistas científicas sincronizan su publicación a la vez. Esto es lo que ha ocurrido con este nuevo atlas celular y sus cuatro artículos correspondientes.
En el primer estudio, se presenta la Tabula Sapiens, un atlas celular que aporta 400 tipos de células diferentes obtenidos de 24 órganos. Para ensamblar los datos, se ha secuenciado el ARN de 500.000 células vivas de distintas personas, analizando las que venían de un solo donante y haciendo comparaciones cruzadas controladas por sus antecedentes genéticos, edad, exposición ambiental y efectos epigenéticos.
El segundo atlas se ha hecho a partir de más de 200.000 células congeladas. Las muestras proceden de 8 órganos humanos sanos de 16 donantes. Además, se han empleado técnicas de aprendizaje automático para asociar las células del atlas con miles de enfermedades relacionadas con un solo gen, así como dolencias relativas a factores genéticos complejos. El objetivo es descubrir qué tipos de células y qué genes están involucrados en ciertas enfermedades.
El sistema inmunitario humano se ha abordado en el tercer estudio, que ofrece el atlas celular del sistema inmunitario, no solo de células sanguíneas, sino de las células que están localizadas en el resto de los tejidos. Para ello, los autores de este equipo de investigación han secuenciado células inmunitarias adultas y en desarrollo de 16 tejidos de 12 donantes adultos, obteniendo la expresión génica de más de 300.000 células. Además, han desarrollado una herramienta de inteligencia artificial llamada CellTypist con la que han identificado 101 tipos celulares distintos, bien por sus características o por su estado, analizando más de un millón de células.Por último, el cuarto artículo, se ha centrado en 9 tejidos prenatales. Así, se ha elaborado un atlas unicelular y espacial del sistema inmunitario en todas las etapas de gestación. Este trabajo revela que el desarrollo de las células sanguíneas y las del sistema inmune no solo se produce en los órganos hematopoyéticos primarios sino en muchos tejidos periféricos.
Del transcriptoma a la información espacial
Como estos atlas celulares se basan en información genómica, no todos ellos incluyen información sobre la morfología de las células que se analizan, es decir, no siempre vemos cómo son esas células o dónde se sitúan. Para ello, es preciso combinar otros métodos, como los análisis histológicos o los mapas celulares de alta resolución.
“En nuestro estudio sobre el desarrollo del sistema inmunológico humano sí hemos incluido una imagen de campo claro bajo la información transcriptómica, por lo que tenemos la información morfológica de las células y del microambiente del tejido en el que se encuentran”, ha afirmado a Diario Médico Sarah Teichmann, directora de Genética Celular del Instituto Wellcome Sanger y copresidenta del comité organizador del Atlas Celular Humano. “La resolución es de 50 micras, por lo que no se trata de una sola célula precisamente. El incluir otras tecnologías de mapeo está definitivamente en el radar de la comunidad científica”, ha asegurado.
Para Stephen Quake, presidente de CZ Biohub Network y catedrático de bioingeniería y de física aplicada de la Universidad de Stanford, aún se puede hacer más. “Muchos de nuestros tejidos los han analizado patólogos capacitados. Tenemos tinciones e imágenes ópticas que proporcionan una base para empezar a conectar la morfológica con el perfil de expresión génica. Además, solo usamos una pequeña fracción de todos los tejidos para hacer el trabajo, por lo que podemos volver atrás y revisarlos”, ha comentado Quake a este diario.
“En los atlas celulares tenemos dos brazos, el brazo celular y el brazo espacial”, ha explicado Aviv Regev, copresidenta del Atlas Celular Humano y catedrática Instituto Broad y del MIT. “El plan es tener ambos totalmente representados. También quiero destacar la importancia de los algoritmos de aprendizaje profundo, que nos ayudan a combinar un mismo tipo de datos de diferentes tejidos y son excelentes para identificar si lo que parecen manzanas o naranjas, en realidad, son la misma cosa”, ha añadido.
Secuenciada por primera vez la ‘zona gris’ del genoma humano
Un estudio en organoides muestra que los diabéticos son más susceptibles al SARS-CoV-2
Así identifica el cerebro que hemos cometido un error y aprendemos
Uno de los pioneros y mayores expertos del mundo en la técnica de secuenciación de una única célula dirige varios de los proyectos para la construcción del atlas celular humano. Se trata de Kun Zhang, catedrático del Departamento de Bioingeniería de la Universidad de California, San Diego (UCSD). Zhang ha participado en el encuentro Age Open Science que se ha celebrado esta semana en Salamanca y al que ha acudido Diario Médico.
Allí ha comparado el esfuerzo científico en obtener el atlas celular humano con la secuenciación de nuestro genoma. “Hace veinte años, cuando se elaboró el gran proyecto del genoma humano, hubo varios grupos que trabajaron solo en uno o dos cromosomas, como el cromosoma 21 o el 13. Cuando lo juntaron todo, se obtuvo el gran genoma humano”, ha explicado a este medio.
Aunque, en esta ocasión, Zhang no forma parte del macroestudio publicado este jueves en Science, sí lo ha hecho con anterioridad. “Esta investigación la coordina un gran consorcio para el que mi grupo ha contribuido con el atlas de varios órganos humanos: el atlas del cerebro, del pulmón y del riñón”, ha señalado Zhang. “Las publicaciones que salen ahora van a tener un significado importante», ha afirmado. “Serán como cuando se trabajó en uno de esos cromosomas hace 20 años, cuando se trataba de publicar el genoma humano”.
El de la especie humana no es solo el único atlas que se ha construido. Los hay de muchos otros organismos, de distintos órganos o tejidos. El de primates no humanos (macacos) se ha publicado recientemente en la revista Nature. En él han participado 35 instituciones internacionales. Entre ellas está el grupo del CIBER de Enfermedades Neurodegenerativas liderado por Pura Muñoz, la Premio Nacional de Investigación Ramón y Cajal de Biología 2021, profesora de Biología Celular ICREA de la Universidad Pompeu Fabra (UPF) e investigadora del laboratorio de regeneración tisular del Centro Nacional de Investigaciones Cardiovasculares (CNIC).
Células senescentes
Muñoz, que no ha participado en el atlas celular humano, es experta en los procesos de envejecimiento y, gracias a la técnica de scRNA-seq ha identificado distintos tipos de células senescentes, como ha expuesto en el encuentro de Salamanca. Desde allí, ha valorado el estudio en humanos para este diario y ha comentado la repercusión que los atlas celulares tendrá en el estudio de enfermedades relacionadas con la edad.
“Las células senescentes forman parte de los tejidos cuando envejecemos. También están presentes siempre que haya un daño”, ha comentado. El proceso de daño tisular conlleva la aparición de estas células, “que algunas veces son beneficiosas, pero, casi siempre son perjudiciales”, ha indicado. Lo que ocurre es que, al envejecer, comienzan a fallar los mecanismos que se encargan de eliminarlas y, con el paso del tiempo, “ejercen un papel negativo sobre las células de su entorno. Son unos invitados que causan problemas cuando hay daño y el problema es que aumentan durante el envejecimiento”, ha asegurado Muñoz.
En cuanto a las técnicas de secuenciación de célula única, tanto del primate como del ser humano “vamos a ver que aumenta el número de células dañadas, que no son buenas, en los tejidos”, ha destacado Muñoz. “Estas publicaciones nos dicen cómo estamos cuando envejecemos para poder ver las causas del envejecimiento y ver qué podemos hacer para revertirlo. Si no conocemos bien cómo estamos, no podemos hacer nada para luchar contra ello”, ha añadido.
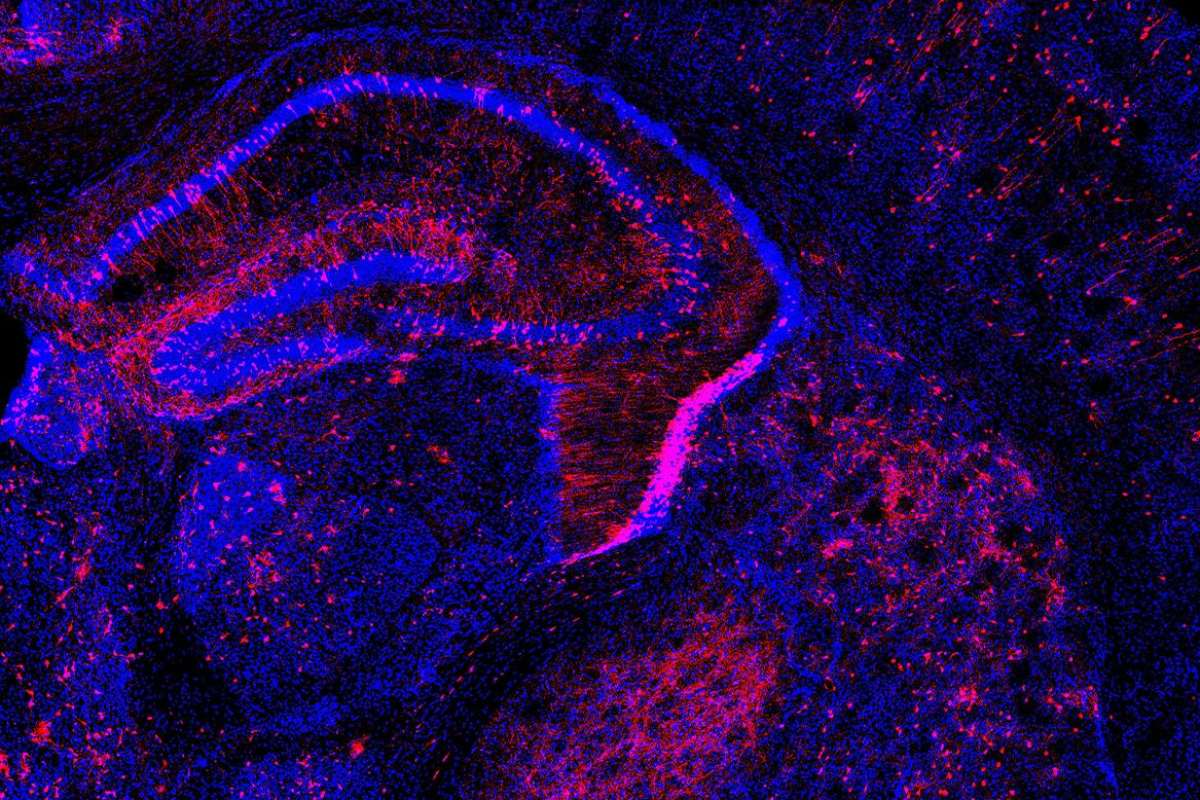 El método ‘Pertub-seq’ permite introducir cambios, como en esta célula de cerebro murino. Foto: LABORATORIO P. ARLOTTA (HARVARD)
El método ‘Pertub-seq’ permite introducir cambios, como en esta célula de cerebro murino. Foto: LABORATORIO P. ARLOTTA (HARVARD)
 Se cree que el Homo erectus fue el primer homínido en conocer el fuego.
Se cree que el Homo erectus fue el primer homínido en conocer el fuego.
 El Homo erectus es un buen candidato a ser la especie previa al Homo sapiens.
El Homo erectus es un buen candidato a ser la especie previa al Homo sapiens. Muchos Homo erectus pueden haber convivido con los primeros Homo sapiens.
Muchos Homo erectus pueden haber convivido con los primeros Homo sapiens.
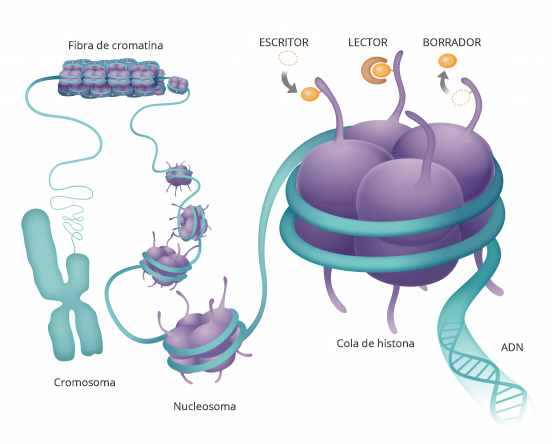
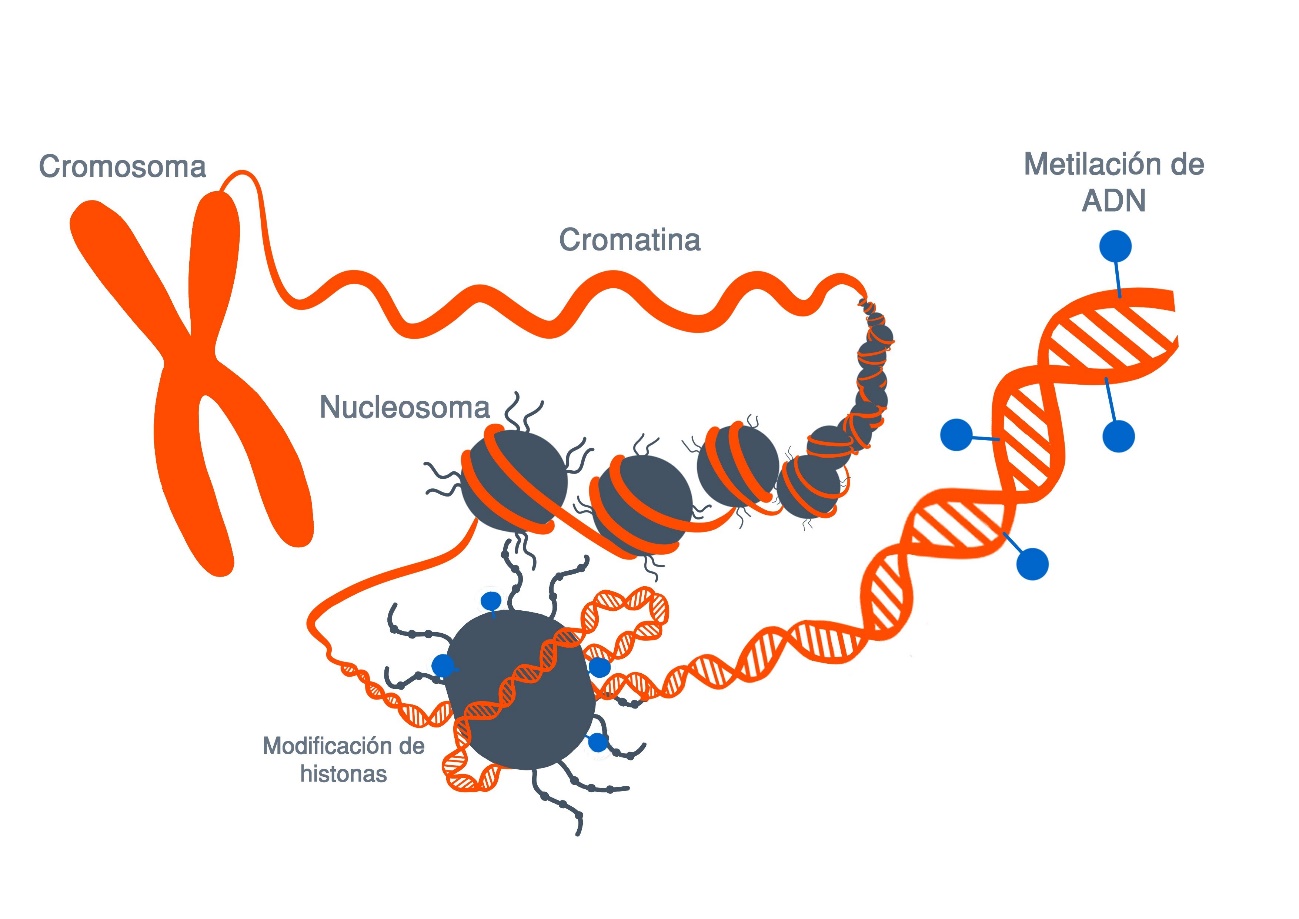
 Metilacion en el dolor
Metilacion en el dolor
 Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas
Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas
Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas

 Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas
Células individuales organizadas por su presencia en órganos y sistemas de todo el cuerpo humano. Foto: Aviv Regev y Anna Hupalowska, Human Cell Atlas Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas
Integración computacional de perfiles de una sola célula y genética para identificar genes de enfermedades y tipos de células en todo el cuerpo humano. Foto: Aviv Regev and Anna Hupalowska, Human Cell Atlas








 El drago milenario de Icod de los Vinos, en la isla de Tenerife.MIGUEL ÁNGEL GARCÍA / FLICKR.
El drago milenario de Icod de los Vinos, en la isla de Tenerife.MIGUEL ÁNGEL GARCÍA / FLICKR.
 Floresta .
Floresta . Foto:
Foto:  Foto:
Foto:  Foto:
Foto:  Foto:
Foto:  Un ejemplar de Pinus longaeva en la Sierra de la Serpiente
Un ejemplar de Pinus longaeva en la Sierra de la Serpiente
 Foto:
Foto: 
 Foto:
Foto: 

